Evoluzione e filogenesi
Enciclopedia della Scienza e della Tecnica (2007)
Evoluzione e filogenesi
Tra la biologia evoluzionistica e la sistematica biologica c'è stato, fin dai tempi di Jean-Baptiste Lamarck (1744-1829) e di Charles Darwin (1809-1882), uno stretto rapporto di reciproca illuminazione. Nell'opera di Lamarck, per esempio, i principi evoluzionistici discussi nella Philosophie zoologique del 1809 hanno un'immediata ricaduta sull'architettura che il naturalista francese darà negli anni seguenti alla sua monumentale Histoire naturelle des animaux sans vertèbres (1815-22). In questa sorta di enciclopedia zoologica, la trattazione si dispiega ‒ ribaltando una secolare tradizione che vedeva l'enumerazione delle specie animali muovere da quelle 'superiori' verso quelle 'inferiori' ‒ attraverso livelli organizzativi successivi, a partire dalle forme più semplici verso quelle più complesse. C'è da osservare, peraltro, che in quest'opera non vi è l'idea di una comune derivazione di tutti i viventi da un unico antenato, in quel variegato rapporto di parentele che oggi siamo soliti rappresentare attraverso la metafora dell'albero. Ogni specie, invece, percorrerebbe un proprio cammino evolutivo indipendente da quello compiuto dalle altre specie e la maggiore o minore complessità di ciascuna di esse e la loro diversa autonomia dall'ambiente sarebbero solo una testimonianza del percorso più o meno lungo tracciato dalla singola linea evolutiva nel corso delle sue trasformazioni nel tempo.
L'idea di un common descent di tutte le specie, cioè dell'universale derivazione da un'antichissima forma di vita primordiale, trova invece spazio in On the origin of species di Darwin (1859). Il capitolo XIII di quest'opera raccoglie dalla classificazione degli animali e delle piante una serie di argomenti a sostegno della visione evoluzionistica e questa, di lì a poco, animerà lo sforzo di Ernest Haeckel, inteso proprio a ricostruire, in chiave di divenire storico delle forme, le relazioni fra i diversi gruppi già riconosciuti dai sistematici. Centrale, in quest'opera, sarà l'adozione di quelli che verranno chiamati 'alberi filogenetici': soluzioni grafiche in cui l'albero si offre insieme come modello geometrico per una rappresentazione di relazioni e come pregnante e forse pericolosa metafora di un processo di progressiva trasformazione, dal lontano passato fino al presente e, insieme, dal semplice al complesso.
Inevitabilmente, forse, l'uomo continua per molto tempo a rappresentare il termine di paragone privilegiato, rispetto al quale i diversi gruppi dei viventi vengono valutati per riconoscere il loro 'grado di avanzamento' rispetto alle forme di vita più primitive. Ciò equivale, però, a ignorare l'eguale distanza che separa da queste ultime, sulla scala del tempo, tutti i viventi attuali, semplici o complessi che siano. Va poi considerata la natura arbitraria dei criteri in base ai quali si viene appunto a riconoscere a un determinato organismo vivente una precisa posizione in termini di complessità strutturale: vana è stata, per esempio, l'illusione di potersi fondare sulle proprietà del genoma, visto che il numero di geni presenti nel nucleo di una cellula umana è largamente inferiore a quello di molte piante o di animali che si è facilmente tentati di considerare più semplici dei Vertebrati da un punto di vista anatomico e funzionale.
Lettura di un albero filogenetico
Fino a epoca recente, gli alberi filogenetici ‒ disegnati in maniera naturalistica, con tutte le nodosità del tronco e il capriccioso portamento dei rami, o ridotti all'essenziale linearità della pura rappresentazione geometrica ‒ nascondevano una pericolosa ambiguità. Essi infatti venivano trattati il più delle volte alla stregua degli alberi genealogici, lungo i rami dei quali si leggevano i rapporti antenato → discendente, a partire da un remoto capostipite. Allo stesso modo, negli alberi filogenetici si volevano leggere relazioni di discendenza, del tipo 'gli Uccelli derivano dai Rettili', 'i Vertebrati a quattro zampe derivano dai Pesci', o 'l'uomo discende dalle scimmie' (e pare già un segno di provata cultura scientifica l'aver evitato la più popolare, e ancor più erronea espressione, secondo la quale 'l'uomo discende dalla scimmia', come se di scimmie ne esistesse una sola specie).
Ben diversa, invece, dovrebbe essere la lettura di un albero filogenetico, come è stato finalmente chiarito nella seconda metà del Novecento dalla moderna sistematica cladistica. In un albero filogenetico, le specie sono i fiori singoli (o le foglie isolate) che si trovano all'estremità di ciascun ramo, ma non occupano mai i nodi, cioè i punti di ramificazione. È lecito invece immaginare questi nodi come altrettanti antenati ipotetici, dei quali possiamo in parte ricostruire le caratteristiche, ma essi non possono essere identificati con specie reali, viventi o estinte che siano. L'intera struttura dell'albero, in effetti, è solo intesa a esprimere le relazioni di maggiore o minore prossimità dell'ultimo antenato in comune che una determinata specie A ha avuto nei confronti di ciascuna altra specie (B, C, D, ecc.) presa in considerazione.
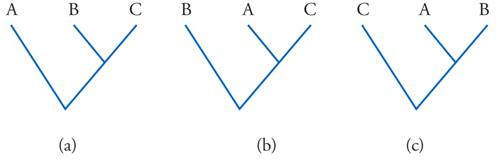
Il concetto può essere meglio illustrato prendendo in considerazione il più semplice fra i possibili problemi di filogenesi, quello che riguarda le reciproche relazioni fra tre specie, che chiameremo A, B e C. Assumeremo per semplicità, come in effetti si è soliti fare, che tutti i punti di ramificazione dell'albero filogenetico siano delle dicotomie. Pertanto, per le relazioni filogenetiche nell'ambito del complesso di specie A, B, C, si possono ipotizzare tre diverse topologie (fig. 2): delle tre specie, quelle che condividono tra loro un antenato più recente, rispetto al più remoto antenato che entrambe hanno in comune con la terza specie, potranno essere, rispettivamente A e B, oppure A e C, oppure B e C. Con l'aumentare del numero delle specie prese in considerazione, il numero dei possibili alberi filogenetici cresce in maniera vertiginosa. Questo è vero, naturalmente, sul piano teorico, perché in realtà uno solo, fra questi alberi, corrisponde effettivamente alla storia evolutiva del gruppo di specie in questione.
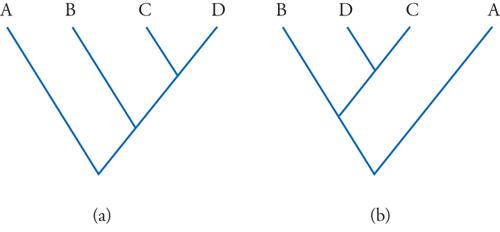
Obiettivo principale della moderna sistematica filogenetica o cladistica è appunto quello di ricostruire l'albero che con maggiore probabilità ripropone le reali relazioni di parentela fra le specie considerate. In un albero filogenetico possiamo per esempio leggere che, fra tutti gli animali diversi dall'uomo, la nostra specie ha il suo più stretto parente nello scimpanzé (per essere più precisi, nel genere che comprende lo scimpanzé comune e lo scimpanzé nano o bonobo) e che il parente immediatamente più remoto è il gorilla, seguito poi dall'orangutan e, quindi, dai gibboni. Ma tutto questo non significa affatto che l'uomo discenda dallo scimpanzé, né che il gorilla abbia dato origine a quest'ultimo e all'uomo, e così di seguito. Queste ultime affermazioni sono, semplicemente, senza senso. Ma è importante sottolineare anche che un albero filogenetico non ci autorizza nemmeno a dire che i gibboni sono scimmie antropomorfe inferiori, dal momento che il loro grado di parentela con l'uomo è molto più remoto, rispetto alla vicinanza che con la nostra specie hanno il gorilla e, soprattutto, gli scimpanzé. Allo stesso modo, nessun albero filogenetico ci autorizza a considerare i Pesci, gli Anfibi o i Rettili come Vertebrati inferiori, in contrapposizione con gli Uccelli e i Mammiferi, che sarebbero invece i Vertebrati superiori. Più in generale, gli alberi filogenetici non offrono alcuna giustificazione per la qualifica di 'inferiore' o 'superiore' che si volesse attribuire a una qualsiasi specie, o a un gruppo di specie. Se crediamo di poter riconoscere posizioni alte e posizioni basse in un albero filogenetico, ciò dipende solo dalle arbitrarie soluzioni grafiche che abbiamo scelto di adottare per rappresentarli (fig. 3).
Sarebbe del resto inopportuno affermare che una distinzione tra forme inferiori e forme superiori trova la sua giustificazione proprio in una lettura storica, evoluzionistica, delle differenze tra i viventi. Nella misura in cui le loro linee evolutive sono sopravvissute fino ai nostri giorni, dimostrando la loro idoneità ai rispettivi ambienti, un batterio, un lombrico o una farfalla 'valgono' né più né meno dell'uomo. Ciò non significa che le ovvie differenze strutturali tra l'uno e l'altro debbano essere ignorate o che non siano il frutto di una lunga storia evolutiva. Ma non sono i loro rapporti filogenetici ad autorizzarci a disporli lungo una serie ideale che va dal semplice al complesso. Questa serie nasce da un confronto dei loro piani organizzativi che prescinde del tutto dalla storia e che, di fatto, ha trovato la sua più esplicita espressione proprio in un contesto pre-evoluzionistico. Un contesto dove la metafora dominante non è quella dell'albero, bensì quella della scala a pioli, lungo la quale i viventi possono essere disposti in un ordine lineare (comunque arbitrario) di crescente complessità. È la metafora della scala naturae che Charles Bonnet (1720-1793) adotta nella Contemplation de la nature (1764). Purtroppo, la tendenza a leggere gli alberi filogenetici quasi fossero alberi genealogici ha incoraggiato il perdurare di un linguaggio che la lettura evoluzionistica dei rapporti fra i viventi avrebbe dovuto da tempo cancellare.
Filogenesi reticolata
La filogenesi è l'insieme dei rapporti di parentela fra i viventi, rapporti che vengono correntemente espressi attraverso rappresentazioni grafiche ad albero. Va osservato, però, che quest'ultima metafora si dimostra, in alcuni casi, inadeguata. Vi sono stati in effetti, nella storia della vita, eventi in cui due o più linee evolutive, anziché rimanere separate ed eventualmente andare incontro, ciascuna per proprio conto, a successive ramificazioni, si sono unite tra loro, dando origine a organismi di origine composita (simbiotica), a partire dai quali è poi ripreso il consueto ciclo di suddivisioni dicotomiche. Eventi di questo tipo costituiscono fasi a evoluzione reticolata, che non si possono ovviamente rappresentare con la tradizionale topologia ad albero. Il più importante fra questi eventi di confluenza tra più linee evolutive è il complesso di eventi che ha dato origine alla prima cellula eucariotica di tipo moderno.
Quello che oggi è il mitocondrio, organulo cellulare perfettamente integrato nella vita della compagine cellulare eucariotica ma che ancora conserva un suo genoma autonomo, ancorché ridotto, era in origine un batterio a vita libera, con una lunga storia evolutiva separata da quella dell'altro unicellulare, del quale appunto sarebbe diventato un organulo, e che ora domina la compagine cellulare con la ricchezza di informazioni contenuta nel suo genoma nucleare. Un batterio fotosintetico a vita libera era anche l'antenato del cloroplasto delle piante, anch'esso ormai ridotto a organulo cellulare, che tuttavia conserva un piccolo genoma proprio. A questi antichi episodi di evoluzione reticolata si possono avvicinare quelli, numerosi, che in epoca più recente hanno dato origine alle simbiosi licheniche tra linee evolutive diverse di funghi e di alghe. In tutti questi casi, ogni tentativo di ricostruire una filogenesi secondo una topologia ad albero porterebbe di necessità a risultati falsi.
Il metodo comparativo e la nozione di omologia
La ricostruzione dei rapporti filogenetici che intercorrono fra tre o più specie è un'operazione semplice dal punto di vista concettuale, ma spesso impegnativa sul piano pratico, e non sempre si conclude con risultati destinati a essere subito accettati senza ulteriori approfondimenti. Essa rappresenta l'aspetto più importante, ai nostri giorni, di una branca della biologia le cui radici affondano in un lontano passato storico. Dobbiamo infatti guardare a quella disciplina che, nata in un contesto pre-evoluzionistico come anatomia comparata, merita attualmente il nome più comprensivo di 'biologia comparata', dal momento che l'ambito delle sue applicazioni si è esteso ben oltre il confine della sola morfologia. Se vogliamo legare simbolicamente a un nome e a una data la nascita di questa disciplina, l'anatomia comparata ha preso le mosse da un'opera del 1555, l'Histoire de la nature des oyseaux di Pierre Belon (1517-1564). In essa troviamo infatti un preciso e articolato confronto fra uno scheletro umano e uno scheletro d'uccello. L'operazione è compiuta con l'ausilio di due figure tra loro affiancate, in cui le singole ossa dell'uno e dell'altro scheletro vengono contraddistinte con lettere dell'alfabeto, curando di utilizzare la stessa lettera, per l'uomo e per l'uccello, quando l'autore abbia motivo di ritenere equivalenti i due pezzi scheletrici. Questo esercizio, del tutto originale all'epoca in cui fu eseguito e che risulta singolarmente corretto nella sua effettiva realizzazione, poteva consentire al lettore di avvicinarsi all'anatomia degli Uccelli prendendo a riferimento l'anatomia di una specie, quella umana, che di certo gli doveva essere maggiormente nota.
Duecentocinquanta anni dopo, questo tipo di comparazione diventa lo strumento quotidiano di analisi utilizzato dai grandi anatomisti della scuola francese di Georges Cuvier (1769-1832) ed Etienne Geoffroy Saint-Hilaire (1772-1844). Quest'ultimo arriva a concepire il principio dell'unità generale di piano strutturale di tutti gli animali, principio che lo spingerà a cercare corrispondenze strutturali anche fra animali dall'organizzazione molto diversa tra loro, come Artropodi e Vertebrati, o Vertebrati e Molluschi. Emergono in ogni caso, dalle ricerche di questi studiosi e dei loro colleghi e allievi presso il Musée d'Histoire Naturelle di Parigi, due nozioni che nel 1843 il paleontologo e anatomista inglese Richard Owen (1804-1892) codifica sotto i nomi di omologia e analogia. Omologie sono proprio le corrispondenze che Belon veniva sottolineando, quando riconosceva le stesse ossa in due organi così diversi fra loro, sia per forma che per funzione, come l'arto superiore dell'uomo e l'ala dell'uccello. Analogie sono invece le somiglianze, dettate in genere dalla comune risposta alle stesse esigenze funzionali, che esistono fra parti anatomiche grossolanamente simili, ma che possono essere costruite anche con elementi anatomici differenti, come nel caso delle ali degli Insetti e degli Uccelli, ovvero in quello delle pinne di una foca, di un pinguino e di uno squalo.
La biologia comparata era giunta a questi risultati a prescindere da ogni interpretazione storica delle forme viventi, ma è chiaro che questo complesso mosaico di somiglianze interpretabili, di volta in volta, come omologie o come analogie si presta benissimo a una lettura in termini evoluzionistici. Non a caso Darwin vi dedica mezzo capitolo di On the origin of species. Se le analogie appaiono ora come il frutto di simili pressioni selettive esercitate su organismi appartenenti a linee evolutive diverse (così che, per es., piante di numerose famiglie hanno assunto, in ambienti estremamente aridi, un simile aspetto di pianta succulenta), le omologie sono interpretate, in chiave evoluzionistica, come precise testimonianze di parentela. In effetti, che cosa se non la comune derivazione da uno stesso antenato potrebbe spiegare la presenza di una stessa impalcatura scheletrica, con omero, radio, ulna, carpali, metacarpali e falangi, in appendici corporee così diverse come la zampa anteriore di una lucertola o di un cane, l'arto superiore di un uomo, l'ala di un pipistrello o di un uccello, la pinna di una balena, o l'arto scavatore di una talpa?
Dalla definizione di Owen, secondo il quale omologo sarebbe "lo stesso organo sotto ogni varietà di forma e funzione", si passa pertanto a una nozione storica di omologia, definita come "somiglianza strutturale dovuta alla derivazione da uno stesso antenato". Per essere più precisi e parafrasando una definizione di Walter Bock, diremo che una struttura X presente in un organismo A e una struttura Y presente in un organismo B sono omologhe se derivano, storicamente, da una medesima struttura Z presente nell'ultimo antenato comune condiviso da A e B. Il termine critico, in quest'ultima definizione, è l'aggettivo 'ultimo', cioè il più recente. Il valore di questa precisazione appare chiaro non appena ci si chieda se l'ala di un uccello e l'ala di un pipistrello siano strutture omologhe. Posta in questi termini, senza ulteriori specificazioni, la domanda è ambigua. Da un lato, infatti, è facile dimostrare che l'ultimo antenato comune a Uccelli e pipistrelli non possedeva ali. Si trattava certamente di un vertebrato quadrupede, che appoggiava al suolo tutte e quattro le zampe, così come i primi Rettili (dai quali, tra l'altro, sono discesi gli Uccelli) e i primi Mammiferi. La trasformazione dell'arto anteriore in un organo atto a sostenere il corpo nell'aria è un'innovazione occorsa indipendentemente lungo le due linee evolutive che portano, rispettivamente, agli Uccelli e ai pipistrelli.
Ne è la riprova il fatto che la superficie portante è realizzata, nei due casi, con soluzioni del tutto diverse: un ventaglio di penne in un caso, una membrana tesa fra le dita della mano nell'altro. D'altro lato, però, è ben vero che l'impalcatura scheletrica delle due ali è sostanzialmente la stessa, le differenze più evidenti riguardando il numero e lo sviluppo delle ossa della mano. In entrambi i casi, l'appendice si inserisce sul tronco in corrispondenza di un cinto scapolare, mediante un unico osso (l'omero), al quale fa seguito una coppia di ossa appaiate (radio e ulna), a loro volta seguita dalle ossa carpali e metacarpali e infine dalle falangi. Non v'è dubbio sul fatto che questo piano strutturale, comune all'ala dell'uccello e a quella del pipistrello, sia riconducibile a una simile struttura presente in un antenato comune sia agli Uccelli sia ai pipistrelli. Pertanto, secondo l'uso tradizionale, ottocentesco, del termine, le due strutture sono omologhe. Ma sono omologhe come arti anteriori di un vertebrato a quattro zampe, e non come ali. L'antenato comune al quale occorre risalire per trovare la prima comparsa di questo tipo di appendici non è l'ultimo antenato comune ai soli Uccelli e pipistrelli, bensì l'ultimo antenato comune a tutti i Vertebrati terrestri. Questo carattere in comune, pertanto, ci dà informazioni sulle relazioni filogenetiche all'interno dei Vertebrati, ma solo e precisamente a livello del nodo (punto di ramificazione) in cui esso compare come innovazione dell'ultimo antenato comune a tutti i Vertebrati terrestri, e solo a questi; sui rapporti fra Uccelli e pipistrelli, questo carattere non ci fornisce alcuna informazione.
La ricostruzione della filogenesi
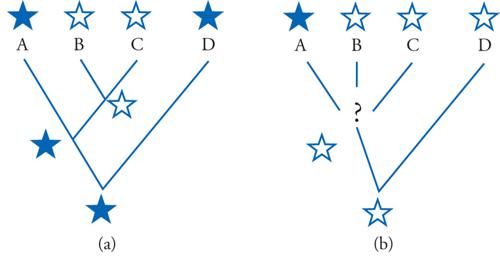
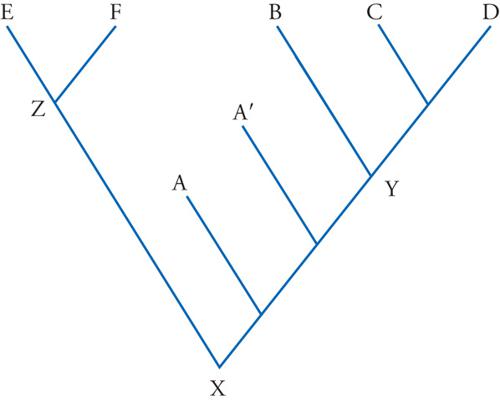
Fu l'entomologo tedesco Willi Hennig (1913-1976) a puntare per primo l'attenzione su questa importante distinzione fra caratteri omologhi riconducibili precisamente all'ultimo antenato comune alle specie oggetto di studio, e solo a esse, e caratteri omologhi riconducibili a nodi più basali, cioè più antichi, dell'albero filogenetico. Hennig propose il nome di 'apomorfie' per i primi e di 'plesiomorfie' per i secondi. Dato che solo le apomorfie condivise (sinapomorfie) sono informative in termini filogenetici, diventa criticamente importante la disponibilità di un metodo che permetta di riconoscere se un'omologia condivisa da due specie sia, o meno, una sinapomorfia. Numerosi sono i metodi proposti per risolvere questo problema; ci limiteremo qui a presentare il metodo dell'outgroup comparison. Esso è basato sul principio di parsimonia, secondo il quale la ricostruzione filogenetica più plausibile è quella che presuppone, da parte dalle diverse linee evolutive coinvolte, il più basso numero complessivo di cambiamenti nei caratteri presi in considerazione. Molte analisi filogenetiche correnti prendono in esame, contemporaneamente, decine o anche centinaia di specie, ma per illustrare il concetto sarà sufficiente (e opportuno) prendere in considerazione la più elementare delle ricerche filogenetiche, quella che ha per obiettivo la ricostruzione della filogenesi di sole tre specie, che chiameremo A, B e C. Più numerose sono le specie oggetto di studio (il cd. ingroup), più alto è il numero di caratteri che è opportuno prendere in esame. Anche per tre sole specie, tuttavia, non è detto ci si possa fidare dei risultati suggeriti da un singolo carattere, anche se questo, come nell'esempio che segue, può risultare di per sé informativo sul piano filogenetico. In ogni caso, per semplicità di esposizione ci si limita qui a considerare un solo carattere.
Siano date dunque le tre specie animali A, B e C e si consideri il numero di paia di zampe presente in ciascuna di esse. Se questo numero è uguale in tutte e tre le specie (per es., due paia), il carattere è ovviamente privo di interesse ai fini della ricostruzione dei rapporti filogenetici all'interno del nostro ingroup. La situazione non sarebbe migliore se ciascuna specie presentasse una condizione diversa dalle altre, per esempio due paia di zampe nella specie A, un solo paio nella specie B e nessuna zampa nella specie C. Il carattere potrebbe invece apparire informativo (ma non lo è necessariamente) nel caso in cui solo una delle tre specie differisse dalle altre due, avendosi, per esempio, due paia di zampe in A e in C e un solo paio in B. Non v'è dubbio che in una sistematica biologica tradizionale questa distribuzione di caratteri avrebbe facilmente indotto a raggruppare tra loro A e C in uno stesso genere, o in una stessa famiglia, a cui si contrapporrebbe un genere, o una famiglia, ospitante la sola specie C. Tuttavia, se il nostro scopo è quello di riconoscere i rapporti filogenetici all'interno delle tre specie, non possiamo procedere ulteriormente fino a che non si abbia una ragionevole stima della natura (plesiomorfa o apomorfa) del carattere condiviso da A e C. In altre parole, in questo gruppo ci sono stati passaggi evolutivi da una condizione a quattro zampe verso una condizione a due zampe, o cambiamenti in senso opposto, o addirittura in entrambe le direzioni?
Per affrontare questo problema servendosi del metodo dell'outgroup comparison (fig. 4) è necessario prendere in considerazione uno o più outgroup. Per outgroup si intende una specie esterna al complesso di specie oggetto del nostro problema filogenetico (cioè all'ingroup): in altre parole, essa deve essere (verosimilmente almeno) imparentata con le specie dell'ingroup più alla lontana di quanto due qualsiasi di queste siano imparentate fra loro. Leggendo queste relazioni su un albero filogenetico, troveremo dunque che l'ultimo antenato in comune fra l'outgroup e il complesso delle specie dell'ingroup corrisponde a un nodo più basale rispetto ai nodi che esprimono le relazioni filogenetiche all'interno dell'ingroup. L'outgroup, tuttavia, dovrà essere sufficientemente vicino all'ingroup da consentire estese comparazioni: sul piano morfologico almeno, un lombrico o una mosca non sarebbero outgroup adeguati per valutare i rapporti filogenetici di un ingroup costituito da uomo, aquila, alligatore. Tornando all'esempio precedente, si andrà dunque a verificare se nell'outgroup, che chiameremo D, siano presenti due paia di zampe, come in A e in C, o un paio solo, come in B. È facile dimostrare che, per il principio di parsimonia, la condizione presente nell'outgroup è la più antica (plesiomorfa) fra le due alternative presenti nell'ingroup.
Si daranno, pertanto, due casi. Se nell'outgroup D è presente un solo paio di zampe, come in B, la condizione opposta, condivisa da A e C, rappresenta una sinapomorfia di questi ultimi e suggerisce quindi che le due specie condividano tra loro un antenato comune più recente (in corrispondenza del quale sarebbe avvenuta la transizione dalla condizione bipede a quella quadrupede), non condiviso invece da B, che si raccorderebbe al gruppo (A, C) solo a livello di un nodo più profondo nell'albero filogenetico. Se, invece, nell'outgroup D sono presenti due paia di zampe, come in A e in C, ne risulta che queste due specie sono accomunate da una simplesiomorfia, che nulla dice specificamente a riguardo delle loro parentele. Quanto ai loro rapporti con B, rimarrebbero infatti aperte tutte e tre le possibilità: un antenato comune ad A e B con esclusione di C, un antenato comune a B e C con esclusione di A, e anche una separazione profonda tra la linea evolutiva di B e quella di A e C. Pertanto, se il carattere in comune tra A e C si è rivelato essere una sinapomorfia, l'analisi filogenetica si conclude con la formulazione di un'ipotesi (albero filogenetico), che si può ritenere valida fino a che l'esame di altri caratteri non venga a mettere in dubbio la sua validità; se, invece, il carattere comune ad A e C si dimostra essere una plesiomorfia condivisa (simplesiomorfia), esso dev'essere semplicemente scartato, in quanto privo di informazione in rapporto allo specifico problema affrontato, e si dovranno prendere in considerazione caratteri diversi.
Situazioni di conflitto fra le relazioni filogenetiche suggerite da un carattere, o da un insieme di caratteri, e quelle suggerite da un diverso carattere, o da un diverso insieme di caratteri, sono una regola con ben rare eccezioni. Il problema principale è costituito dall'omoplasia, cioè dalla possibilità che due caratteri apparentemente omologhi siano in realtà dovuti a convergenza (cioè all'indipendente acquisizione di una stessa novità evolutiva da parte di due linee evolutive affini ma distinte), oppure a un cambiamento evolutivo 'a ritroso', che riporta a una condizione non distinguibile da una precedente plesiomorfia (fenomeno, quest'ultimo, assai frequente nel caso di caratteri molecolari). Per superare queste difficoltà sono stati messi a punto diversi metodi matematici intesi a ricavare, da un insieme di dati di per sé contraddittori, le ipotesi dotate di più elevata verosimiglianza. È doveroso ammettere, peraltro, che molte analisi risultano di fatto inconclusive. Un notevole sforzo viene pertanto indirizzato alla ricerca di nuovi caratteri potenzialmente informativi, nonché alla valutazione comparativa dei pregi e dei difetti che classi diverse di caratteri possono manifestare, in relazione al loro impiego nelle analisi filogenetiche.
Ricostruzioni filogenetiche su base molecolare
Il metodo comparativo in biologia ha dunque avuto origine nell'ambito dell'anatomia comparata. Con l'avvento dei moderni metodi cladistici di ricostruzione della filogenesi, i caratteri morfologici hanno continuato per qualche tempo a dominare nelle analisi, ma accanto a essi ha trovato sempre più largo impiego l'utilizzazione di caratteri di natura molecolare. Si tratta, quasi sempre, di segmenti più o meno estesi o completi delle sequenze di nucleotidi che costituiscono le molecole degli RNA o dei DNA, o delle sequenze di amminoacidi che costituiscono le proteine. I progressi tecnologici che a partire dagli anni Ottanta del XX sec. hanno consentito di ottenere sequenze nucleotidiche con crescente efficienza e rapidità e a costi sempre più bassi sono in larga misura responsabili del crescente favore che i dati molecolari hanno trovato nelle analisi filogenetiche, spesso a netto discapito dei dati morfologici. Vi sono, in effetti, ricercatori che ritengono praticamente oziose la raccolta e l'analisi di questi ultimi, in quanto il loro contributo alla ricostruzione della filogenesi sarebbe trascurabile, se non addirittura fuorviante. Spesso, tuttavia, accanto ai dati molecolari continuano a essere considerati anche quelli di natura morfologica. Alcuni ricercatori ritengono preferibile analizzare separatamente le due classi di dati, riservando a un momento successivo il confronto tra le ricostruzioni filogenetiche separatamente ottenute. Altri ritengono invece preferibile inserire le une e le altre in una stessa matrice di dati, la quale viene assoggettata a un'unica analisi integrata.
Molti alberi filogenetici sono stati costruiti sulla base delle sequenze nucleotidiche di un singolo gene, ma ci sono buone ragioni per affermare che, in assenza di adeguate informazioni in contrario, tali alberi rappresentano in effetti la filogenesi delle varianti del gene in questione, ma non sempre quella delle specie campionate. Un problema frequente, in proposito, è dato dai fenomeni di duplicazione genica, vale a dire da quegli eventi che portano, all'interno del genoma di un organismo, alla formazione di copie multiple di un gene, ciascuna delle quali può andare incontro a una propria evoluzione indipendente. Di regola, una o più copie rimangono funzionali, altre potranno invece degenerare in pseudogeni. Le une e le altre, comunque, potranno conservarsi per tempi lunghissimi e trasmettersi alle specie che deriveranno dall'antenato in cui è avvenuta la duplicazione. Ne deriva una doppia e complessa serie di relazioni. Da un lato, tra le diverse forme del gene presenti nello stesso organismo, che vengono dette 'paraloghe', esiste una relazione di parentela direttamente riconducibile alla duplicazione genica; dall'altro, in specie viventi tra loro imparentate potranno esistere le varianti omologhe (dette 'ortologhe') di ciascun paralogo o, forse, solo di alcuni fra i paraloghi. È infatti possibile che alcuni di questi, soprattutto se privi di un valore funzionale, tendano a perdersi, in maniera non prevedibile, nell'una o nell'altra linea evolutiva. Pertanto, se a seguito di una duplicazione un gene a si trova a essere rappresentato da due diverse forme paraloghe, che chiameremo a1 e a2, è possibile che alcune specie discendenti le conservino tutte e due, mentre altre potrebbero conservare solo a1, altre ancora solo a2. Un'analisi che non tenga conto di questo fatto, o che non sia in grado di identificare le singole linee paraloghe, può portare pertanto a confronti impropri, per esempio fra il paralogo a1 della specie X con il paralogo a2 della specie Y, erroneamente presi, semplicemente, come le copie del 'gene a' rispettivamente presenti nell'una e nell'altra specie.
Ontogenesi e filogenesi
L'opportunità di guardare alle successive tappe dello sviluppo embrionale degli organismi come a una fonte di informazione da utilizzare a scopo classificatorio è un'idea che precede l'avvento della visione evoluzionistica dei fenomeni biologici. Importanti sono, in proposito, le osservazioni del grande embriologo Karl Ernst von Baer (1792-1876). Nel primo volume (1828) della sua opera Über Entwicklungsgeschichte der Thiere: Beobachtung und Reflektion egli osservava la regolare progressione con cui, nel corso dello sviluppo, si fanno manifesti nell'embrione caratteri progressivamente più specifici: dapprima quelli che permettono la sua attribuzione a una determinata classe zoologica, poi quelli propri del suo particolare ordine di appartenenza, infine quelli diagnostici a livello di genere e di specie.
In un contesto evoluzionistico, non tarda a farsi strada l'idea che la rapida sequenza temporale degli stadi di sviluppo (a partire dai primi stadi embrionali, assai semplici sul piano morfologico, fino al raggiungimento delle complesse strutture dell'adulto) corrisponda, in modo più o meno preciso, alla successione delle tappe evolutive attraverso le quali si è evoluta la corrispondente specie attuale, a partire dai più remoti e semplici antenati unicellulari. È l'idea che Ernst Haeckel (1834-1919) codifica nel suo 'principio biogenetico fondamentale', secondo il quale, appunto, l'ontogenesi (cioè la storia dell'individuo) ricapitola la filogenesi (cioè la storia della specie lungo l'albero dei viventi). Il principio biogenetico di Haeckel avrebbe validità se nel corso dell'evoluzione l'ontogenesi si modificasse sempre e solo per aggiunte terminali, cioè progressivamente spostando verso il segmento embrionale o giovanile dello sviluppo l'architettura corporea già propria dell'adulto, mentre intanto la sequenza delle tappe di sviluppo si allunga, raggiungendosi la maturità solo in corrispondenza di una condizione finale nuova. Ma non è difficile dimostrare che questa è solo una delle molteplici possibilità di cambiamento dell'ontogenesi nel corso della filogenesi.
L'inattendibilità della legge biogenetica è una delle ragioni che hanno portato, per buona parte del Novecento, a un disinteresse generalizzato per la biologia dello sviluppo da parte degli studiosi di biologia evoluzionistica. A tale risultato hanno molto contribuito, peraltro, anche i genetisti della prima metà del secolo, a cominciare da Thomas H. Morgan (1866-1945), con un programma di ricerca centrato soprattutto sui meccanismi di trasmissione dei geni da una generazione all'altra e sulla loro distribuzione nelle popolazioni, piuttosto che sui processi attraverso i quali i geni si realizzano nel fenotipo. In particolare, il ricorso prioritario agli esperimenti di incrocio, quasi per definizione circoscritti all'ambito intraspecifico, non consentiva di affrontare il problema dell'origine delle forme biologiche, che richiede confronti anche tra specie molto distanti, con piani organizzativi assai diversi. Solo a partire dagli ultimi anni Ottanta ha cominciato a prendere consistenza uno sforzo di integrazione fra la biologia evoluzionistica, fortemente legata alla genetica delle popolazioni, e la biologia dello sviluppo. Determinante, da questo punto di vista, è stata l'evoluzione della moderna genetica molecolare dello sviluppo, che ha dimostrato la presenza, in specie appartenenti a gruppi zoologici molto diversi, di un grande numero di geni in comune, coinvolti ‒ negli animali più diversi ‒ nel controllo della formazione delle stesse strutture: per esempio, il gene pax6 per l'occhio, il gene tinman per il cuore. Ne è nata la moderna biologia evoluzionistica dello sviluppo, nell'ambito della quale è riemerso, tra l'altro, anche l'interesse per la comparazione delle sequenze ontogenetiche di animali diversi a fini di ricostruzione filogenetica.
Particolarmente promettenti appaiono, al riguardo, i moderni studi sull'eterocronia. Con questo termine si intendono le differenze di natura temporale che si possono riconoscere in una comparazione fra le sequenze degli stadi di sviluppo di due animali diversi. Questo tipo di confronti si applica, naturalmente, nell'ambito di gruppi zoologici che condividano le principali linee strutturali dell'architettura corporea: per esempio, nell'ambito dei Vertebrati, e ancor meglio all'interno di una singola classe, come i Mammiferi. Le differenze temporali che vanno sotto il nome di 'eterocronie' possono riguardare, per esempio, il momento in cui compare il primo abbozzo di un determinato organo (cuore, cervello, ecc.), ma non è facile individuare una scala temporale normalizzata, alla quale riferire contemporaneamente le sequenze degli eventi ontogenetici di animali diversi. Pertanto, il moderno indirizzo della ricerca sulle eterocronie concentra la propria attenzione sull'ordine in cui, nelle differenti specie, avvengono i diversi eventi ontogenetici. Per esempio, se la comparazione riguarda una serie di animali dei quali si prende in considerazione il momento in cui compaiono i primi abbozzi di cuore (Cu), cervello (Ce), fegato (Fe) e polmoni (Po), una specie potrà essere caratterizzata dalla sequenza temporale CeCuFePo, un'altra dalla sequenza CeFePoCu, e così di seguito. Su queste sequenze può essere quindi compiuta un'analisi filogenetica.
Filogenesi e classificazione
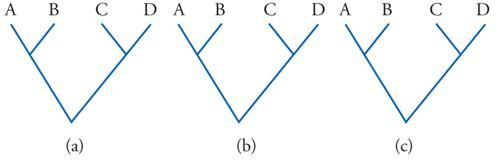
Le conseguenze più evidenti della sistematica applicazione dei moderni metodi di ricostruzione filogenetica riguardano la sistematica biologica. Se accettiamo il principio che la classificazione dei viventi debba essere fondata sui loro rapporti filogenetici (e sembra difficile proporre un principio più ragionevole di questo), dobbiamo essere pronti a rinunciare a molti gruppi tradizionali. Per la sistematica filogenetica (o cladistica), infatti, sono accettabili in una classificazione solo i gruppi definiti da apomorfie, cioè da tutti (e soli) i discendenti da un ipotetico antenato, corrispondente a un nodo più o meno profondo nell'albero filogenetico. Questi gruppi vengono detti 'gruppi monofiletici' (o 'cladi', cioè rami dell'albero). Vanno invece esclusi i gruppi polifiletici e quelli parafiletici (fig. 6). Polifiletici sono i gruppi definiti da omoplasie, i cui membri ‒ per quanto simili possano apparire ‒ appartengono quindi a più linee evolutive e non sono riconducibili a uno specifico antenato comune. I gruppi parafiletici, infine, sono quelli definiti da plesiomorfie. I loro membri, pertanto, sono riconducibili a un antenato comune, ma non rappresentano per intero l'insieme dei discendenti da questo, dal momento che ne viene escluso un sottoinsieme, caratterizzato da apomorfie che il residuo gruppo parafiletico non condivide.
Non vi è, naturalmente, contrasto di opinioni sulla necessità di rimuovere dalla classificazione ogni eventuale gruppo polifiletico che vi fosse rimasto. In zoologia, per esempio, si è perduta memoria del momento in cui fu abbandonato il gruppo polifiletico dei vermi; più recente è lo smembramento della vecchia classe polifiletica dei Pesci. In botanica, il termine alghe viene ormai scritto con l'iniziale minuscola, a indicare un raggruppamento di natura ecologica, e non più una divisione nella classificazione dei vegetali. Più controversa è la situazione a riguardo dei gruppi parafiletici, che non hanno spazio in un coerente sistema filogenetico, ma la cui legittimità, nelle classificazioni, trova ancor oggi non pochi difensori. C'è da ritenere, tuttavia, che queste resistenze si faranno sempre più deboli. Esempi di gruppi parafiletici sono gli invertebrati, i Rettili e le Dicotiledoni. Si tratta, infatti, di gruppi monofiletici incompleti, da ciascuno dei quali è stato escluso un sottogruppo monofiletico ben caratterizzato: rispettivamente, i Vertebrati, gli Uccelli e le Monocotiledoni. In altre parole, gli invertebrati sono i Metazoi (gli animali pluricellulari) meno i Vertebrati, i Rettili sono i Sauropsidi meno gli Uccelli, le Dicotiledoni sono le Angiosperme (le piante a seme) meno le Monocotiledoni.
La biologia comparata contemporanea
A tutt'oggi, il significato della rivoluzione cladistica non è stato ancora ben assimilato dalla biologia, al di fuori dell'ambito specialistico della ricostruzione filogenetica e delle sue conseguenze classificatorie. Nella biologia sperimentale, frequentissimi sono gli studi in cui vengono messi a confronto Vertebrati e invertebrati, come se i due raggruppamenti fossero tra loro equivalenti. Un confronto sensato può essere invece proposto fra i Vertebrati e il loro sister group, cioè il gruppo che dai Vertebrati diverge a livello dell'ultimo nodo che immediatamente precede quello che sta alla base di tutti i Vertebrati. Con molta probabilità, questo gruppo è rappresentato dai Cefalocordati, ai quali appartiene l'anfiosso, un piccolo animale marino, la cui organizzazione e, soprattutto, lo sviluppo embrionale del quale sono stati molto spesso presi in considerazione, fin dalla fine dell'Ottocento, come un utile termine di comparazione con i Vertebrati. Nel caso degli Uccelli, una comparazione sensata sul piano filogenetico non li dovrà contrapporre ai Mammiferi (che con ogni probabilità hanno raggiunto la condizione omeoterma indipendentemente da essi) e nemmeno ai Rettili intesi nel senso tradizionale (cioè tartarughe, tuatara, lucertole, serpenti e coccodrilli), bensì ai soli Coccodrilli, che ne rappresentano il sister group.
L'analisi filogenetica risulta, in effetti, l'indispensabile punto di partenza per ogni studio di tipo comparativo. Si consideri, per esempio, l'affermazione, più volte ripetuta nella letteratura zoologica, secondo la quale i ragni tropicali del genere Nephila sarebbero caratterizzati da maschi nani. In effetti, in questo genere il dimorfismo sessuale è molto pronunciato e le dimensioni delle femmine sono di parecchie volte superiori a quelle dei maschi. Questa situazione, tuttavia, non giustifica di per sé la qualifica di 'nano' attribuita ai maschi. Il legittimo termine di paragone, infatti, non è rappresentato dalle femmine conspecifiche, bensì dai maschi dei generi affini. In parallelo, le gigantesche femmine di Nephila andranno confrontate con le femmine dei generi affini piuttosto che con i loro maschi. Una corretta analisi comparativa potrebbe forse confermare il nanismo dei maschi, ma potrebbe invece concludersi con la constatazione che le dimensioni dei maschi di Nephila non sono significativamente diverse da quanto atteso, in rapporto alla posizione filogenetica del genere; questi ragni potrebbero essere interessati, invece, a un gigantismo delle femmine. Di fatto, un'analisi filogenetica ha dimostrato che in questo genere sono presenti sia il nanismo maschile sia il gigantismo femminile: una ricostruzione che alla fine rende meglio comprensibile il fortissimo dimorfismo sessuale di Nephila.
Un esempio ancor più clamoroso della necessità di interpretare l'evoluzione dei caratteri (siano essi morfologici, molecolari o comportamentali) all'interno di un rigoroso contesto filogenetico è fornito da una serie di generi americani di vespe, che costruiscono nidi di diversa fattura, apparentemente adatti a essere collocati, nell'ambiente, in posizioni diverse. I più sofisticati fra questi nidi sembrano essere quelli di Leiocnemis. Essi sono formati da uno strato di cellette che viene costruito sulla pagina inferiore di grandi foglie, in ambiente forestale. La superficie esterna di questi nidi è verde come il substrato sul quale il nido viene realizzato ed è ornata di costolature in rilievo che hanno l'aspetto di nervature di foglia. Sarebbe facile costruire, al di fuori di ogni analisi filogenetica, uno scenario in cui le vespe più primitive di questa linea evolutiva avrebbero cominciato a costruire nidi sulla pagina inferiore delle foglie, proteggendoli così dalla pioggia e dall'insolazione, oltre che da eventuali nemici che osservino dall'alto. Una serie di 'migliorie' successive avrebbe contemplato l'adozione di una superficie costolata e infine il camuffamento mediante l'adozione di un materiale dello stesso colore del substrato. Ma non è così. Una filogenesi di queste vespe, costruita sulla base di caratteri non comportamentali, ha dimostrato che nell'evoluzione del nido l'ultimo atto è stato proprio la scelta della pagina inferiore delle foglie come sito di nidificazione: i nidi verdi e costolati erano già disponibili, ma venivano realizzati su altro substrato.
Fossili e ricostruzione filogenetica
Di fronte a un albero filogenetico, si può essere tentati di collocare un fossile conosciuto proprio in corrispondenza di un nodo. Per esempio, perché non identificare con Archaeopteryx lithographica l'ipotetico antenato degli Uccelli moderni? Perché non porre Ichthyostega o Acanthostega proprio alla base della radiazione di tutti i moderni Vertebrati a quattro zampe? Questo tipo di identificazione, in effetti, non è raro nella letteratura divulgativa, ma nemmeno quella scientifica ne è del tutto immune. Ma si tratta di un'identificazione fondata su un'errata interpretazione del significato degli alberi filogenetici. Un albero filogenetico non è un albero genealogico, per cui non esprime relazioni antenato → discendente, nemmeno se l'insieme delle forme prese in considerazione include specie di età geologica differente. Naturalmente, in un albero filogenetico c'è spazio anche per le forme estinte, oltre che per quelle viventi. L'incompletezza (che spesso è drammatica frammentarietà) delle informazioni disponibili per gli organismi del passato rende semplicemente più problematica la loro collocazione, ma nell'albero filogenetico c'è spazio per tutti i rami, siano essi estinti o sopravvivano invece fino ai nostri giorni.
Questa circostanza causa però un problema di tipo classificatorio. Prendiamo per esempio il caso degli Uccelli. L'insieme degli Uccelli attuali costituisce indubbiamente un gruppo monofiletico, riconducibile a un nodo che corrisponde all'ultimo antenato comune fra due specie tra loro remote come, per esempio, lo struzzo e l'usignolo. Vi sono Uccelli fossili che appartengono indubbiamente a tale gruppo monofiletico: per esempio, specie estinte di pappagalli, di rapaci notturni, di passeracei. La loro condizione di specie conosciute solo allo stato fossile, e sicuramente estinte, nulla toglie alla possibilità di collocarle all'interno degli Uccelli. Qual è, però, la posizione di Archaeopteryx? È certo che questo vertebrato estinto condivide con gli Uccelli (come sopra definiti) un antenato comune dal quale non sono derivati animali oggi viventi diversi dagli Uccelli, ma è altrettanto certo che Archaeopteryx non discende dall'ultimo antenato comune ai soli Uccelli attuali.
La situazione in cui si trova Archaeopteryx, rispetto al gruppo più prossimo definito sulla base delle specie oggi viventi, è comune a moltissimi fossili, soprattutto fra quelli meno recenti. Per assegnare a queste forme una precisa posizione classificatoria, nel rispetto della filogenesi, si è convenuto di adottare una precisa convenzione: la distinzione fra stem group e crown group. Quest'ultimo è il gruppo monofiletico definito sulla base delle specie viventi, mentre lo stem group è la serie parafiletica di specie (o gruppi di specie) che stanno, come Archaeopteryx rispetto al crown group degli Uccelli, in una posizione intermedia fra il nodo che separa il crown group dal suo più prossimo vicino con rappresentanti viventi e il nodo che sta alla base del crown group stesso (fig. 8). Intorno agli anni Ottanta del Novecento, quando la sistematica filogenetica veniva affinando i suoi metodi, numerosi sono stati gli studiosi secondo i quali l'analisi filogenetica doveva essere condotta esclusivamente sulle forme attuali, salvo poi trovare, negli alberi così ottenuti, una conveniente collocazione per le forme conosciute solo allo stato fossile. Questa posizione è oggi sostenuta solo da un numero esiguo di ricercatori. Nella maggior parte degli studi più recenti i fossili, quando sono disponibili, vengono utilizzati nelle analisi filogenetiche assieme alle forme viventi.
Bibliografia
Hall 1992: Hall, Brian K., Evolutionary developmental biology, London, Chapman & Hall, 1992.
Hennig 1966: Hennig, Willi, Phylogenetic systematics, Urbana, University of Illinois Press, 1966.
Lecointre, Le Guyader 2003: Lecointre, Guillaume - Le Guyader, Hervé, La sistematica della vita, Bologna, Zanichelli, 2003.
Mayr 1999: Mayr, Ernst, Storia del pensiero biologico, Torino, Bollati Boringhieri, 1999.
Minelli 1991: Minelli, Alessandro, Introduzione alla sistematica biologica, Padova, Muzzio, 1991.
Minelli 1993: Minelli, Alessandro, Biological systematics, London, Chapman & Hall, 1993.
Minelli 2003: Minelli, Alessandro, The development of animal form, Cambridge, Cambridge University Press, 2003.
Wägele 2000: Wägele, Johann Wolfgang, Grundlagen der phylogenetischen Systematik, München, Pfeil, 2000.
Wiley 1981: Wiley, Edward O., Phylogenetics, New York, Wiley, 1981.
© Istituto della Enciclopedia Italiana - Riproduzione riservata