DNA antico
Frontiere della Vita (1998)
DNA antico
A partire dalla seconda metà degli anni Ottanta, l'impiego della tecnica della reazione a catena della polimerasi (PCR, Polymerase Chain Reaction) ha fornito agli studiosi di paleontologia e archeologia molecolare un potente strumento per studiare il DNA antico: già numerose sono le pubblicazioni sul DNA estratto da una vasta gamma di organismi estinti, dai microbi ai vertebrati. Gli studiosi stanno ora valutando questi primi risultati per ottenere nuove informazioni sulla sopravvivenza del DNA antico, la sua riproducibilità tramite PCR e le eventuali contaminazioni da parte del DNA esogeno, tutti problemi di grande rilevanza. Anche se ottenere DNA antico autentico ed endogeno da organismi morti da molto tempo è di per sé un'impresa, la disponibilità di marcatori molecolari è importante per confermare precedenti studi basati su metodi analitici tradizionali e per poter acquisire nuove informazioni, altrimenti non ottenibili, sull'identificazione delle specie, sulle loro relazioni filogenetiche e sulla loro storia evolutiva.
Introduzione
Con il termine DNA antico si indica un DNA ottenuto da resti di organismi deceduti, anche se tra gli studiosi non vi è ancora pieno accordo sul lasso di tempo che deve intercorrere tra la morte dell' organismo e il prelievo del suo DNA affrnché quest'ultimo possa essere ascritto alla categoria del DNA antico. Non vi è sostanziale disaccordo sul fatto che il DNA estratto da specie estinte costituisca DNA antico, tuttavia se le specie si stanno estinguendo alla allarmante velocità che si dice, significa che un organismo potrebbe essere morto soltanto da un anno o anche da un solo mese. Per quanto riguarda l'uomo, c'è da chiedersi quali vittime di omicidio possano costituire casi di DNA antico e quando i casi giudiziari debbano essere inclusi in questa categoria. Poiché in questo caso il DNA viene utilizzato per semplici analisi svolte nel corso di un procedimento giudiziario e i cui risultati possono essere utilizzati durante il processo, esiste una differenza con il DNA estratto da specie estinte? Alcuni ricercatori hanno suggerito che la definizione di DNA antico dovrebbe tener conto del grado di deterioramento dell'organismo: solo quando questo si è deteriorato al punto da rendere necessaria l'analisi del DNA per la sua identificazione, allora si può parlare di DNA antico. Tuttavia, bisogna tener conto del fatto che i tempi di deterioramento variano ampiamente, in relazione alle condizioni ambientali, da poche ore a molte settimane. Questi casi comunque riguardano l'ambito giudiziario e non rappresentano casi di DNA antico. Tuttavia, in medicina legale, quando si tratta di analisi da materiale riesumato per la determinazione delle caratteristiche razziali, gli studi molecolari sono considerati come casi rappresentativi di DNA antico. Cosa è, allora, DNA antico?
Certamente ogni DNA estratto da fossili (resti di organismi conservati nella crosta terrestre da tempi preistorici, 10.000 anni o più) o da subfossili (resti più recenti di 10.000 anni) di specie oggi estinte, è DNA antico. l problemi sorgono quando ci si trova di fronte a reperti più recenti. In questo articolo considereremo come DNA antico solo il DNA endogeno estratto da resti di almeno 50 anni. Questa definizione è, per un certo verso, arbitraria e si basa, grosso modo, sul tempo di una generazione umana.
Il campo di studi del DNA antico è relativamente recente e, sebbene vi siano stati convegni annuali sull'argomento e vi sia un gruppo di studiosi dediti a questo tipo di ricerche, solo dal 1996 esiste una rivista scientifica specifica per questo settore, Ancient Biomolecules. Quindi gli studiosi si sono tenuti in contatto tramite notiziari e incontri scientifici periodici. Il primo notiziario dedicato al DNA antico fu pubblicato addirittura due anni prima che apparisse, nel 1984, il primo articolo scientifico sul tema. Nel 1982, J. Tkach, un medico di Bozeman, nel Montana, cultore di paleontologia, radunò attorno a sé un piccolo gruppo di studiosi entusiasti e specializzati in biologia molecolare, tutti interessati allo studio delle prime forme di vita e al DNA. Nel 1983 Tkach diede vita a The extinct DNA newsletter e il gruppo fu chiamato The extinct DNA study group. Lo scopo di questo gruppo di ricerca era, come dichiarato anche nel primo notiziario (febbraio 1983), quello di scambiarsi idee e informazioni su una serie di progetti futuristi tra cui "il recupero di geni da organismi estinti". Due successivi numeri del notiziario furono pubblicati nel marzo e nell'aprile del 1983. L'autore di questo articolo è uno dei primi membri del gruppo e può testimoniare l'entusiasmo e la serietà delle discussioni che vi si tenevano e ricorderà per sempre i particolari della prima conferenza su tale tema, organizzata da Tkach il 9 marzo del 1983.
Circa dieci anni più tardi, nell'aprile del 1992, è apparso il primo numero di Ancient DNA Newsletter, come mezzo di comunicazione tra gli studiosi impegnati nel recupero di frammenti di genoma da animali estinti. Il notiziario era redatto da R. Wayne e da A. Cooper e il suo scopo era quello di rendere noti "miglioramenti nelle procedure di laboratorio e nell'analisi di dati importanti per l'estrazione e la caratterizzazione del DNA proveniente da materiale antico". Il secondo e il terzo numero di Ancient DNA Newsletter sono stati pubblicati, rispettivamente, nel dicembre del 1992 e nel febbraio del 1994; da allora sono state pubblicate diverse centinaia di articoli scientifici riguardanti il DNA antico. Lo scopo di questo articolo è quello di illustrare i principali risultati ottenuti finora in questa disciplina emergente e di discutere alcuni dei principali problemi incontrati dai ricercatori.
Problemi incontrati nella ricerca sul DNA antico
Numerosi problemi assillano gli studiosi del DNA antico a causa della natura dei materiali in esame e della varietà di condizioni a cui tali materiali sono stati sottoposti. l problemi principali saranno discussi qui di seguito.
Piccole quantità di DNA
In condizioni normali, non appena un organismo muore, il suo DNA inizia a degradarsi. Il tasso di degradazione e la quantità di DNA che si degrada dipendono dal lasso di tempo che intercorre tra la morte e il momento dell'analisi e dalla tafonomia dell'organismo, intendendo con tale termine l'insieme delle condizioni ambientali e dei processi cui è sottoposto l'organismo dopo la morte, inclusi i processi di fossilizzazione. L'impossibilità di recuperare DNA dalla maggior parte dei fossili e subfossili è imputabile principalmente a modificazioni dovute a processi autolitici e a contaminazioni associate alle varie condizioni ambientali in cui si sono trovati tali reperti. Pertanto, anche quando è possibile recuperare DNA da un organismo antico, questo si rinviene in piccole quantità e deve essere amplificato prima di poter essere studiato; tale amplificazione veniva inizialmente effettuata mediante il clonaggio in batteri.
L'invenzione della PCR da parte di K. Mullis si rivelò di grande utilità per la ricerca nel campo del DNA antico: invece di dover clonare frammenti di DNA, questi potevano essere amplificati direttamente tramite l'analisi delle sequenze. Divenne altresì possibile utilizzare oligonucleotidi specifici per selezionare e amplificare determinate regioni dello stampo fossile: ciò rese possibile ottenere numerose copie di un frammento di DNA antico in tempi relativamente brevi. Per la maggior parte dei campioni di DNA antico, la lunghezza dei frammenti di DNA mitocondriale amplificati è generalmente compresa tra 100 e 200 bp (base pairs, coppie di basi), fino a un massimo di circa 500 bp ottenuta da campioni di pelle particolarmente ben conservati, provenienti da musei (Pääbo, 1990). Alcuni autori hanno suggerito che l'amplificazione di frammenti di DNA antico superiori alle 500 bp debba essere verificata attentamente per le possibili contaminazioni da DNA esogeno. Comunque, oggi è divenuto possibile amplificare vari brevi frammenti sovrapposti gli uni agli altri e ricostruire così lunghe sequenze unendo questi brevi frammenti di amplificazione. Ciò è stato fatto per ricostruire le sequenze di rDNA mitocondriale del bradipo di terra estinto (Höss et al., 1996).
DNA antico danneggiato
L'idrolisi, seguita da depurinazione, cioè il distacco delle basi puriniche, e frammentazione, è il primo danno a cui va incontro il DNA antico (Lindahl, 1993a).
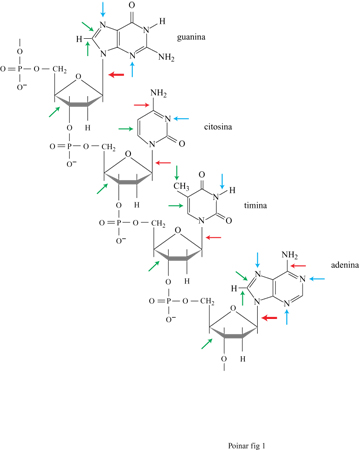
Oltre alla depurinazione del DNA, i tessuti antichi subiscono un' alterazione dovuta ai processi ossidativi (fig. 1) che modificano soprattutto le pirimidine e i residui degli zuccheri, e determinano la formazione di siti privi di base nucleotidica e di legami crociati intramolecolari (Pääbo, 1989). Da uno studio relativo a materiali secchi provenienti da tessuti molli, di età compresa tra i 4 e i 13 mila anni, è emerso che i campioni più antichi possiedono concentrazioni di guanina e adenina paragonabili a quelle presenti nei tessuti viventi; invece, le concentrazioni di citosina e timina sono ampiamente ridotte, con la seconda che risulta meno del 5% rispetto alla quantità attesa. Inoltre, è stato visto che le pirimidine, specialmente la timina, sono più sensibili al danno ossidativo rispetto alle purine, e che almeno 1 pirimidina su 10 risulta modificata, con l'anello aperto o saturo. L'età del campione di per sé non dà indicazioni assolute sul deterioramento del DNA. Gli effetti ambientali sono di grande importanza e il tasso di frammentazione del DNA dipende dalla velocità di essiccamento dell'organismo dopo la morte. In seguito alla disidratazione dei tessuti, i processi idrolitici endogeni, responsabili della degradazione del DNA, si interrompono o vengono ampiamente ridotti e gli effetti dell'ossidazione risultano minimizzati. Tale fenomeno può spiegare perché insetti e piante inclusi nell'ambra possano preservare il DNA per periodi molto lunghi (milioni di anni): l'umidità degli organismi può essere infatti rapidamente assorbita dai componenti della resina degli alberi (Poinar, 1994).
La lettura di sequenze alleliche su DNA danneggiato può : comportare ulteriori problemi, specialmente quando un'imperfetta copiatura da parte della polimerasi durante la peR introduce errori casuali nelle sequenze. È stato messo in evidenza (Brown e Brown, 1994), che errori di sequenziamento del DNA antico causati dalla presenza di nucleotidi danneggiati nella sequenza stampo sono più frequenti rispetto a quelli del DNA contemporaneo. La ricostruzione degli alleli originali richiede quindi l'analisi di molti cloni, il cui numero varia in relazione all'entità del danno; si devono esaminare almeno 20 o più cloni per una sequenza di 200 bp.
Problemi di contaminazione
Le condizioni di laboratorio necessarie per la ricerca sul DNA antico devono essere più rigorose rispetto a quelle impiegate in altri settori della biologia molecolare, al fine di evitare possibili contaminazioni. L'estrazione del DNA deve essere effettuata in una camera sterile, poiché è in questa fase che si verificano la maggior parte delle contaminazioni. Tuttavia, elementi inquinanti possono essere presenti nell'organismo stesso prima della sua morte o, successivamente, penetrarvi in qualunque momento, incluso quello in cui si effettua l'analisi delle sequenze.
La maggior parte delle contaminazioni non derivanti dall'intervento del ricercatore si verificano subito dopo la morte dell' organismo e sono imputabili a fonti sia esterne che interne. Le contaminazioni interne partono, generalmente, dall'apparato gastrointestinale e da quello urogenitale; i batteri e i funghi che sono presenti in questi apparati, così come i microbi introdotti con gli alimenti, possono moltiplicarsi e invadere i tessuti del cadavere. La maggior parte dei contaminanti esterni deriva, invece, da insetti e da altri decompositori attratti dal cadavere (Smith, 1986). Il tasso di invasione dei tessuti corporei da parte di contaminanti esterni e interni (per lo più batteri e funghi) dipende comunque dall'ambiente e dalla localizzazione del cadavere. M. Höss e collaboratori (1996) nella loro ricerca sulla filogenesi molecolare dei bradipi di terra estinti, hanno constatato che solo un millesimo del DNA amplificabile derivava da quello dei tessuti del bradipo, mentre il resto apparteneva a contaminanti, in gran parte batteri e funghi.
L'inquinamento del campione può avvenire, però, anche durante la sua raccolta; questo tipo di contaminazione è generalmente imputabile a DNA umano, anche se vi possono essere altre sorgenti contaminanti da parte degli stessi ricercatori che derivano, per esempio, dagli abiti indossati o da precedenti contatti verificatisi con animali o piante; inoltre, la contaminazione può avvenire anche durante la conservazione del campione, specialmente nei musei, dove cuti e pelli sono conservati nelle medesime teche, spesso in presenza di artropodi saprofagi. lnfine, la contaminazione da parte di DNA esogeno può avvenire durante il processo di preparazione ed estrazione del DNA dal campione. A tale proposito, nel corso dei diversi convegni sul DNA antico, sono state suggerite alcune norme generali (Handt et al., 1994; Stoneking, 1995): l) il laboratorio deve essere caratterizzato da condizioni di estrema pulizia e utilizzato per la sola estrazione di DNA antico; questo tipo di laboratorio deve essere dotato di filtri per la circolazione dell'aria, finestre sigillate, irradiazione continua tramite raggi UV, sterilizzazione quotidiana dei banchi di lavoro con varechina (questa è da preferirsi perché la maggior parte degli altri agenti sterilizzanti non è in grado di distruggere le spore batteriche); inoltre illaboratorio deve essere il più possibile isolato (da piani o, se possibile, situato in edifici separati) da altri laboratori dove siano conservati tessuti o DNA di specie dello stesso gruppo di quelle in studio; qualora, infine, il DNA antico in questione sia di origine umana, sono necessarie ulteriori precauzioni; 2) le manipolazioni successive all'estrazione devono essere eseguite in un laboratorio sterile separato; devono essere condotti controlli regolari su possibili contaminazioni dell'acqua (è questa una fonte di contaminazione molto più rilevante di quanto, normalmente, non si ritenga); tutte le soluzioni tampone devono essere filtrate, sterilizzate in auto clave e controllate su piastre di crescita per l'eventuale presenza di altri organismi (naturalmente così non si potranno evidenziare cellule epiteliali ma i batteri che a esse sono frequentemente associati); 3) si devono effettuare controlli periodici dei reagenti del laboratorio per possibili contaminazioni, mediante PCR su campioni scelti in modo casuale; 4) devono essere rimossi i contaminanti da tutta la superficie del campione, sia asportando fisicamente gli strati più esterni, nel caso si tratti di elementi scheletrici, sia sterilizzando la superficie stessa del campione (tutte le persone che lavorano con il DNA antico devono aver seguito dei corsi sulle tecniche di sterilizzazione). In passato queste nozioni costituivano parte integrante dei corsi di microbiologia generale e, agli studenti si insegnava a sterilizzare le superfici e gli strumenti di laboratorio e a determinare la presenza di contaminanti. Oggi i raggi UV, gli abiti sterili, l'acqua quattro volte distillata e altri strumenti moderni ci hanno reso meno consapevoli della necessità di adottare tecniche di sterilizzazione. Comunque, nonostante l'estrema sensibilità delle attuali metodologie che consentono di rilevare anche la presenza di contaminanti cellulari che non possono crescere su semplici piastre di agar, batteri e funghi costituiscono ancora un'importante sorgente di contaminazione da DNA esogeno.
Tempi di conservazione del DNA antico
Questo aspetto, di fondamentale importanza per gli studi sul DNA antico, è stato oggetto di numerose indagini e T. Lindahl (1993a), probabilmente, ha svolto la ricerca più approfondita a tale proposito. Quest'autore ha messo in evidenza come il DNA, similmente alle altre macromolecole biologiche, sia sottoposto a decomposizione spontanea. Tuttavia, mentre i fattori che inducono la decomposizione (idrolisi, ossidazione e metilazione non enzimatica) agiscono sia in vivo che in vitro, le endonucleasi, dedicate a riparare costantemente i danni, agiscono solo in vivo.
Dopo la morte dell'organismo, il DNA completamente idratato si degrada spontaneamente, soprattutto per depurinazione, in piccoli frammenti. Secondo Lindahl (1993a), la degradazione si verifica nel corso di diverse migliaia di anni a temperature moderate, ma può essere ridotta di 5 ÷ 10 volte se il campione viene a trovarsi in condizioni di forza ionica molto alta. Inoltre, il DNA dei tessuti ossei adsorbito a idrossiapatite mostra una riduzione del 50% del tasso di depurinazione. In tal modo, condizioni favorevoli di conservazione, quali ambiente secco e freddo, possono consentire la conservazione del DNA per decine di migliaia di anni. Tali stime sulla longevità del DNA antico sono testimoniate dal recupero di DNA endogeno da tessuti ossei, pelle e materiali vegetali di almeno 50.000 anni, un tempo di sopravvivenza del DNA antico pari a quello stimato possibile da Lindahl (1993a). Eccezioni a questa regola sono rappresentate da materiale biologico protetto dal naturale decadimento ambientale come nel caso di organismi inclusi nella resina fossilizzata o negli strati di rocce sedimentarie; tali esempi atipici saranno discussi nell'ultima parte di questo saggio.
Valutazione della presenza di DNA in campioni antichi
Come è possibile determinare la presenza di DNA recuperabile in un tipico campione di materiale antico? Non è pensabile analizzare l'ultrastruttura di tutti i campioni per determinarvi la presenza di nuclei intatti e, comunque, il DNA potrebbe essere adeso a varie superfici minerali del campione. D'altro canto, l'estrazione di DNA, la sua amplificazione e il suo sequenziamento per tutti i campioni può costituire un inutile dispendio di denaro e tempo. Chiari indizi sul grado di conservazione dei tessuti sono lo stato di integrità esterno e l'ambiente in cui il campione è stato ritrovato. Per molti casi la storia tafonomica completa è ignota e non è possibile stimare le condizioni dei tessuti interni. Purtuttavia sarebbe preferibile poter determinare le probabilità che ha il campione di contenere DNA endogeno. Oggi è divenuto possibile utilizzare il tasso di racemizzazione degli amminoacidi delle proteine come indicatore della presenza di DNA recuperabile da fossili e subfossili. J.L. Bada e collaboratori (1994) furono i primi a suggerire che, a pH neutro, l'energia di attivazione per la depurinazione del DNA nativo è paragonabile a quella necessaria alla racemizzazione degli amminoacidi. Molti amminoacidi sono presenti nella forma di due isomeri ottici, una forma L e una D (enantiomeri). Negli organismi viventi, solo la forma L viene utilizzata per la sintesi delle proteine; quando però un organismo muore, gli amminoacidi L vengono racemizzati nella forma D, fino a raggiungere un rapporto di 1:1.
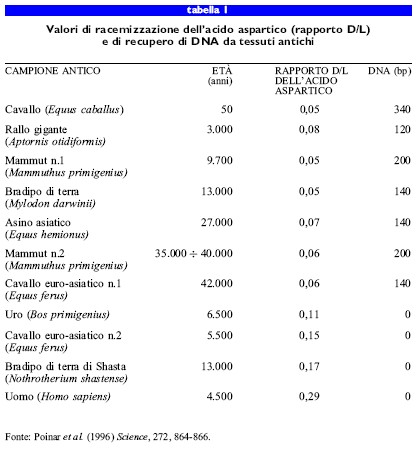
Bada e collaboratori (1994) e X. Wang e collaboratori (1996) hanno applicato questo metodo a insetti conservati in resina fossilizzata e, attualmente, questo stesso metodo viene utilizzato per determinare la probabilità di rinvenire DNA non degradato nei fossili (Poinar et al., 1996). Supponendo che il tasso di racemizzazione dipenda da fattori simili a quelli che determinano la degenerazione del DNA, gli autori hanno utilizzato la racemizzazione dell'acido aspartico come indice della conservazione del DNA. Esperimenti condotti su diversi campioni antichi, di età compresa tra le decine e i milioni di anni, hanno dimostrato che non è possibile ottenere DNA endogeno da campioni in cui il rapporto D/L dell'acido aspartico sia superiore a 0,08.
Il vantaggio pratico di questa correlazione è che, essendo la determinazione del tasso di racemizzazione degli amminoacidi molto semplice e rapida, questa permette di ottenere una stima della probabilità di riuscire a ottenere DNA endogeno dal campione. Un aspetto importante, come si può osservare nella tabella (tab. I), è che, mentre esiste una relazione inversa tra DNA antico e racemizzazione dell'acido aspartico, non esiste una correlazione diretta tra l'età del campione e il tasso di racemizzazione o la quantità di DNA recuperato; ciò è un'ulteriore dimostrazione dell'importanza dei fattori tafonomici esterni e interni per la conservazione del DNA antico.
Studi condotti su vertebrati estinti
Questa parte del saggio illustra alcuni studi condotti su campioni estratti da vertebrati estinti, databili tra gli 80 e i 50.000 anni. Tali studi possono fornire importanti informazioni per l'identificazione delle specie, sulle loro relazioni con taxa ancora esistenti e sulla bio geografia del passato. Qui di seguito sono illustrati i casi più significativi.
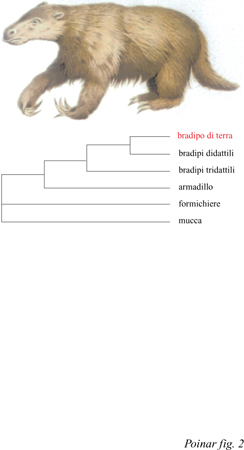
Il bradipo di terra estinto. - Quando Charles Darwin sbarcò nel settembre del 1832 a Bahia BIanca nella provincia di Buenos Aires, in Argentina orientale, non aveva alcuna idea di quello che avrebbe trovato. Appena lui e i suoi accompagnatori ebbero percorso 20 miglia a sud-est, lungo la costa verso Punta Alta, si imbatterono casualmente in un terreno di argilla rossa con inclusioni di rocce calcaree che costituiscono parte della formazione pampeana. Immaginate lo stupore di Darwin quando notò i resti di tre giganteschi bradipi di terra estinti inclusi nel terreno. Uno di essi catturò la sua attenzione in modo particolare, tanto che, successivamente, il professor R. Owen del British Museum lo classificò sulla base dei reperti riportati da Darwin e lo chiamò Mylodon darwinii (fig. 2) dal nome del suo scopritore. Sulla base della posizione e della natura dei resti, Darwin suppose che questi dovessero risalire al 'tardo periodo terziario'. Darwin dedusse che l'animale doveva essere provvisto di una lunga lingua estensibile simile a quella delle giraffe e, da un'analisi accurata della dentatura, Owen concluse che M. darwinii doveva essere vegetariano. Fu quindi ipotizzato che questo bradipo estinto fosse in grado di elevarsi, poggiando sugli arti posteriori, per poi spingere verso il basso i rami degli alberi e mangiarne le foglie. L'osservazione delle caratteristiche strutturali di Mylodon consentì di dedurre le sue abitudini di vita. Questa creatura gigantesca possiede qualche relazione di parentela con i bradipi attuali, di piccole dimensioni e fortemente adattati alla vita arborea? E, in caso di risposta affermativa, a quale dei due generi di bradipi è maggiormente affine? Gli studi sul DNA antico hanno consentito di rispondere a queste domande circa 160 anni dopo il rinvenimento da parte di Darwin. In una recente pubblicazione, M. Höss e collaboratori (1996) hanno riportato i risultati dell'estrazione di DNA da due campioni di tessuto osseo vecchio di 13.000 anni, appartenente a M. darwinii, rinvenuti nella Caverna del Mylodon, a Ultima Esperanza, in Cile. Gli autori hanno tentato di isolare DNA da ossa, tessuti molli, denti e da residui di feci di Mylodon, ma solo il tessuto osseo ha dato risultati positivi, anche se gran parte del DNA estratto derivava da contaminazioni batteriche e fungine. Tuttavia c'era sufficiente materiale endogeno da permettere di sequenziare, in tutto, circa 1000 bp. Gli autori attribuiscono il loro successo al clima secco subantartico e alla bassa temperatura presente all'interno della caverna. Gli autori si sono chiesti se Mylodon sia maggiormente affine, dal punto di vista filogenetico, alle due specie ancora esistenti del genere Choloepus, che comprende i bradipi didattili (Megalonychidae), o alle tre specie di bradipi tridattili del genere Bradypus (Bradypodidae). Entrambi i generi sono oggi presenti esclusivamente nella parte settentrionale dell'America Meridionale e in America Centrale. L'albero filogenetico costruito sulla base del DNA antico di Mylodon e del DNA dei due generi di bradipi attuali e di altri Sdentati ha mostrato che Mylodon è più affine ai bradipi didattili che non a quelli tridattili (v. figura 2). l risultati ottenuti hanno anche dimostrato che le due famiglie attuali dei bradipi non sono strettamente legate filogeneticamente e che la vita arborea è stata acquisita almeno due volte nel corso dell' evoluzione dei bradipi ancestrali a vita terrestre. Per correttezza, è necessario sottolineare che Darwin stesso aveva ipotizzato una stretta relazione tra Mylodon e gli attuali bradipi arborei, anche se non era stato in grado di stabilire con quale di essi fosse maggiormente affine.
Il tilacino (lupo marsupiale). - Il caso di Mylodon darwinii non costituisce l'unico esempio trattato da Darwin e le cui conclusioni sono oggi confermate da studi su DNA antico. Nel suo libro L'origine delle specie, Darwin parlò di somiglianza analogica (oggi definita analogia), osservando che, spesso, i caratteri analoghi o adattati vi non rivestono una particolare importanza per la sistematica, poiché animali appartenenti a due linee evolutive separate possono adattarsi alle medesime condizioni e, quindi, evolvere strutture morfologiche simili. Questi caratteri comuni tendono a nascondere le reali origini di questi animali.
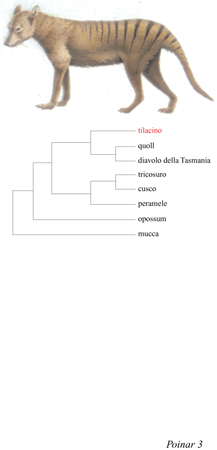
Per esempio, Darwin citò la forte somiglianza tra il cane e il lupo marsupiale australiano, il tilacino (Thylacinus cynocephalus), recentemente estinto (fig. 3). Nonostante vi sia tra loro un'apparente somiglianza, Darwin, lavorando solamente su cranio e ossa, evidenziò grandi differenze nella struttura della dentatura: il cane possiede quattro premolari e due molari per ogni lato dell'arcata mandibolare superiore mentre il lupo marsupiale ha tre premolari e quattro molari; inoltre, vi è una notevole differenza per quanto riguarda la grandezza e la struttura dei denti. La spiegazione di Darwin fu quella che noi oggi defrniamo evoluzione convergente.
I biologi hanno discusso a lungo sul problema dell'analogia e dell'omologia. Con l'avvento della genetica molecolare sembrava che il problema potesse essere risolto una volta per tutte, poiché l'analisi delle sequenze avrebbe consentito la ricostruzione della storia delle specie. Tuttavia, è divenuto oggi evidente che neanche le sequenze, o almeno alcune di esse, sono in grado di distinguere completamente tra analogia e omologia. In merito al tilacino, recentemente alcuni ricercatori sono stati in grado di estrarre DNA da due frammenti di pelle non conciata e da un frammento di tessuto muscolare vecchi di circa 80 anni. È stata ottenuta una sequenza di 210 bp di DNA mitocondriale, per sequenziamento diretto mediante PCR; tale sequenza è stata in seguito confrontata con il DNA corrispondente proveniente da sei generi di marsupiali ancora esistenti dell'America Meridionale e dell'Australia. I risultati ottenuti hanno dimostrato che il tilacino è più affine agli altri carnivori australiani che non ai marsupiali sudamericani e gli autori convengono con l'ipotesi di Darwin di un' evoluzione morfologica convergente del tilacino sia con i carnivori marsupiali dell'America Meridionale che con i veri cani placentati e i lupi (v. figura 3). Analisi successive hanno poi dimostrato che il tilacino è molto diversificato rispetto agli altri marsupiali carnivori australiani e costituisce una linea evolutiva che si è separata molto presto dagli altri membri della famiglia dasiuridi (Krajewski et al., 1992).
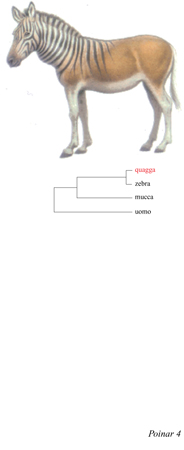
Il quagga sudafricano estinto. - Darwin era stato anche affascinato da ciò che definiva variazioni analoghe, semplici variazioni morfologiche e di colore presenti all'interno della specie. Egli aveva osservato che le variazioni all'interno di una specie sono spesso simili a quelle riscontrabili in una specie affine e che, in alcuni casi, questi caratteri possono ripresentarsi nella forma che si trova in quello che lui definisce progenitore. Come esempio, Darwin citò la presenza di macchie e strisce in alcuni rappresentanti del genere Equus. Notò che spesso negli asini, specialmente nell' età subadulta, sono presenti righe trasversali, simili a quelle delle zebre, sugli arti e, in alcuni casi, anche sulle spalle. Questa osservazione affascinò Darwin che catalogò casi di presenza di strisce sugli arti o sulle spalle in cavalli di varie razze, dalla Gran Bretagna alla Cina orientale. Dopo aver raccolto dati su diversi esperimenti di incrocio tra cavalli e asini, Darwin scoprì una tendenza al ricomparire delle strisce in incroci tra specie filogeneticamente distanti (cavalli, asini e zebre sono considerati specie distinte del genere Equus, anche se possono facilmente dar luogo a ibridi, come nel caso del mulo, incrocio tra un cavallo e un'asina). Darwin aveva anche notato che il quagga (fig. 4) era coperto da strisce come una zebra nella porzione anteriore del tronco ma ne era sprovvisto sugli arti (il quagga, ora considerata una specie distinta, E. quagga, era ancora vivente nella prima metà del 19° secolo, al tempo delle osservazioni di Darwin). Quando il quagga veniva incrociato con un cavallo, l'ibrido era caratterizzato dalla presenza di strisce anche sulle zampe. Sapere se il quagga avesse una stretta relazione con le zebre o con i cavalli non interessava particolarmente Darwin, poiché, come risulta dai suoi scritti, egli assunse che dovesse essere strettamente imparentato alle zebre, benché in grado di incrociarsi con un cavallo.
Quando R. Higuchi e collaboratori (1984) riferirono del recupero di DNA antico da tessuto muscolare essiccato di un esemplare proveniente da collezioni di museo e vecchio di 140 anni, la scoperta ebbe una grande eco, non solo perché costituiva il primo esempio di estrazione di DNA antico ma anche perché veniva confermata l'ipotesi di Darwin: le sequenze di DNA mitocondriale del quagga mostrarono come questo sia strettamente affine alle zebre (v. figura 4).
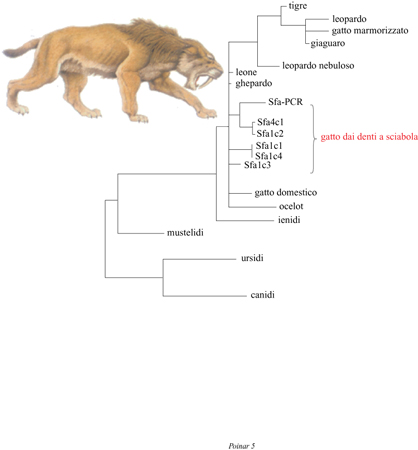
Ulteriori casi di vertebrati estinti. - Il lavoro di D.N.Janczewski e collaboratori (1992) riveste una particolare importanza, poiché riporta l'estrazione di DNA da tessuti ossei conservati in uno strato di catrame. Tale studio include l'analisi di DNA estratto da ossa vecchie di 14.000 anni appartenenti al gatto dai denti a sciabola, Smilodon fatalis (fig. 5). Il confronto del DNA fossile con regioni omologhe di 15 carnivori esistenti ha confermato che il gatto dai denti a sciabola appartiene alla radiazione moderna dei Felidi. Gli autori sostengono che le condizioni di anaerobiosi dell'asfalto hanno costituito un ambiente chimicamente inerte che ha prevenuto la degradazione del DNA e, contemporaneamente, lo sviluppo di microbi che avrebbero potuto decomporre il tessuto. Depositi fossiliferi in catrame come quelli di Rancho La Brea, nell'area di Los Angeles, costituiscono certamente un'ottima fonte di materiale osseo antico. Come fanno notare gli autori, il deposito di La Brea contiene i resti di almeno 59 specie di mammiferi. Fidando nella fortuna, i ricercatori si sono proposti di identificare agenti infettivi, come virus endogeni o altri patogeni che possono aver avuto un ruolo importante nella distribuzione geografica delle specie di mammiferi.
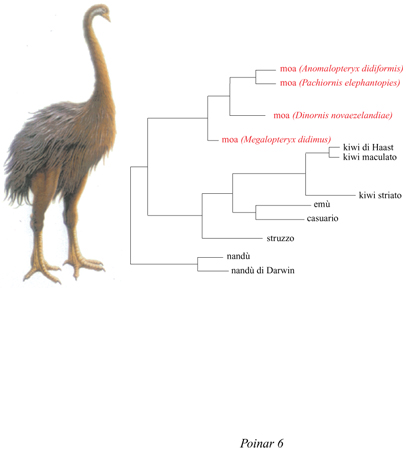
Lo studio del DNA dei moa e il suo confronto con quello degli uccelli Ratiti viventi mostra la possibilità di utilizzare l'analisi del DNA antico per risolvere i problemi di origine e di biogeografia (Cooper et al., 1992). Gli autori hanno estratto e sequenziato circa 400 bp del gene mitocondriale per l'rRNA 12S provenienti da tessuti ossei e molli di quattro specie estinte di moa e di otto Ratiti esistenti (fig. 6). Molti autori ritenevano che il kiwi fosse imparentato ai moa e che la sua taglia ridotta e l'essere insettivoro e notturno ne avessero impedito l'estinzione rapida a opera dei primi polinesiani che hanno colonizzato la Nuova Zelanda. Invece, analisi filo genetiche hanno dimostrato che kiwi e moa hanno origini indipendenti: i primi appartengono a un gruppo australiano-asiatico di Ratiti, assieme all'emù e al casuario, i secondi sono più affini ai Reidi dell'America Meridionale.
Il DNA più antico che sia stato estratto da un mammifero appartiene al mammut lanoso (Mammuthus primigenius), ritrovato in Siberia (Hagelberg et al., 1994; Höss et al., 1994) e datato, in entrambi gli studi, a circa 50.000 anni fa. In questi lavori è emerso come il mammut sia strettamente affine all'elefante piuttosto che ad altri ungulati e che questi animali giganteschi erano molto diversificati. Non è stato ancora possibile determinare se il mammut sia più affine all'elefante indiano (Elephans) o a quello africano (Loxodonta), anche se, al momento della sua scoperta, fu chiamato Elephans primigenius.
DNA antico da vertebrati vecchi più di un milione di anni
Le prime pubblicazioni che trattavano l'isolamento del DNA da resti di vertebrati risalenti a più di un milione di anni fa sono state accolte con scetticismo, poiché è noto che i processi autodegradativi di questa macromolecola si realizzano nell' arco di decine di migliaia di anni (Lindahl, 1993a, 1993b).
Gli unici studi su DNA di vertebrati risalenti a milioni di anni fa sono relativi ai dinosauri. Nel 1995, la stampa pubblicò la notizia che J. Homer e una sua collaboratrice, M. Schweitzer, avevano isolato il DNA da un osso di Tyrannosaurus rex, risalente a 65 milioni di anni e ritrovato a Hell Creek nel Montana (Richardson, 1994). La matrice ossea era eccezionalmente ben conservata al punto che, al microscopio, erano visibili i piccoli globuli rossi nelle lacune ossee. Da quest'osso fu estratto il DNA ma fu impossibile determinare se fosse effettivamente del dinosauro o se provenisse da contaminanti che ne avevano invaso i tessuti ossei; quindi, tale scoperta non è stata mai ufficialmente documentata.
Il solo studio che documenti il recupero di DNA da un dinosauro e che sia stato formalmente presentato al mondo scientifico, è quello di S. Woodward e collaboratori (1994). Questi ricercatori hanno operato su tessuto osseo che, per l'età stimata (80 milioni di anni) e per la localizzazione del ritrovamento, è stato attribuito a un dinosauro. I ricercatori hanno estratto e sequenziato un frammento di DNA mitocondriale corrispondente a una porzione del gene del citocromo b. Da un'analisi eseguita su 174 bp, una sequenza è risultata unica e differente da tutte quelle finora conosciute. Gli autori considerano che questa sia una prova che il DNA estratto appartenga a un dinosauro; tuttavia S.E. Hedges e M.H. Schweitzer (1995), utilizzando un programma (BLAST) per l'analisi della sequenza di dinosauro presunta, pubblicata da W oodward e collaboratori, hanno mostrato che esistono 87 sequenze simili a quella in esame, tutte relative a mammiferi e, tra queste, quella della balena presenta il maggior grado di somiglianza con il presunto DNA di dinosauro. Inoltre, un'analisi filogenetica compiuta, utilizzando il programma BLAST, sulla sequenza di tutti i tetrapodi ha riscontrato un'alta omologia tra la presunta sequenza di DNA del dinosauro e quella umana; per tale motivo, Hedges e Schweitzer hanno ritenuto possibile che il DNA isolato fosse il risultato di una contaminazione di DNA umano.
Un altro gruppo di ricercatori (Zischler et al., 1995) ha ipotizzato, infine, che la presunta sequenza del citocromo b di dinosauro potrebbe rappresentare una remota integrazione di DNA mitocondriale nel genoma nucleare umano. Essenzialmente, anche loro concordano nel ritenere che il DNA isolato da Woodward e i suoi collaboratori fosse il risultato di una contaminazione umana.
Nel concludere questa discussione occorre ricordare lo studio di G.O. Poinar e collaboratori (1996). Questo gruppo ha compiuto un'analisi della sequenza amminoacidica del citocromo b estratto da quattro ossa di dinosauro: un osso di Tyrannosaurus rex, ritrovato a Hell Creek, nel Montana, simile a quello studiato da Homer e Schweitzer; un reperto proveniente dallo Utah, simile a quello studiato da Woodward e collaboratori (1994) e due, ben conservati, ritrovati nell'Antartico. Questi reperti risalgono a un periodo compreso tra i 65 e i 75 milioni di anni fa e i valori del rapporto D/L per l'acido aspartico sono, rispettivamente, 0,23, 0,21, 0,16 e 0,17. Assumendo un valore uguale o minore a 0,08 come soglia per stabilire la possibilità di recuperare DNA da un reperto, questi risultati suggeriscono che queste ossa di dinosauro, e così molte altre, non contengono più DNA endogeno che sia, in qualche modo, utilizzabile.
Resti umani e ritrovamenti archeologici
Da sempre, le mummie hanno esercitato un grande fascino sugli studiosi per i misteri del passato della vita umana che esse portano con sé. P. Lewin (1967) è riuscito a dimostrare la presenza di nuclei e materiale citoplasmatico nell'epidermide di una mano appartenente a una mummia egiziana del Vll secolo a.C. Circa venti anni dopo, S. Pääbo (1985) riuscì a estrarre DNA da una mummia di bambino di 2400 anni fa. In uno studio successivo concernente altre mummie provenienti dal Perù e dall'Egitto, Pääbo (1986) si rese conto che le parti meglio conservate delle mummie sono costituite dalle regioni del corpo più superficiali e periferiche e che non vi è relazione tra l'età di una mummia e il suo stato di conservazione. Sembra più importante invece, il tempo intercorso tra la morte e la disidratazione: più corto è l'intervallo che intercorre tra i due fenomeni, minori sono i danni microbici e l'autolisi.
Poiché è piuttosto raro trovare tessuti molli in resti umani antichi, i risultati dei gruppi di ricerca di E. Hagelberg (1989) e di S. Horai (1989) hanno suscitato un notevole interesse, in quanto sono stati in grado di estrarre DNA mitocondriale da ossa umane antiche. In un lavoro sull'estrazione di DNA mitocondriale da ossa antiche, Hagelberg e collaboratori (1994) hanno dimostrato come sia piuttosto semplice amplificare una discreta quantità di DNA mitocondriale da ossa e come il DNA si conservi intatto più a lungo nel tessuto osseo che nei tessuti molli. Essendo disponibile una grande quantità di materiale archeologico in tutte le parti del mondo, gli studi su ossa di origine umana potranno permetterci di rispondere a numerosi quesiti riguardanti l'antropologia, come, per esempio, i tassi di evoluzione, la genetica delle popolazioni, l'identificazione razziale e culturale e la biogeografia. Secondo Hagelberg il mantenimento dell'integrità del DNA nelle ossa dipende non tanto dalla loro età quanto dalle condizioni tafonomiche. È ovvio che non si conoscono tutti i fattori microclimatici che agiscono sul materiale biologico morto nel terreno. Sarebbe invece di notevole importanza conoscere i meccanismi che regolano il trasferimento e la conduzione del calore in terreni e in strati sedimentari differenti, l' effetto delle variazioni di temperatura diurna e annuale sulle biomolecole presenti nell'ambiente, l'effetto del tipo di terreno in relazione alla capacità di trattenere l'acqua nei materiali antichi e altri aspetti del microclima (Geiger, 1959). In genere, le grotte, in particolare quelle localizzate in aree fredde o temperate, rappresentano il luogo ideale dove reperire ossa e tessuti antichi ben conservati. Le grotte, infatti, normalmente possiedono un'unica apertura verso l'esterno attraverso cui, durante la stagione invernale, entra aria fredda che raggiunge il fondo ove permane a lungo. Tali grotte sono veri e propri magazzini freddi e rappresentano zone privilegiate per la conservazione di macromolecole nelle ossa e in altri tessuti. Queste considerazioni possono spiegare il fatto che la maggior parte del DNA antico recuperato proviene da animali pleistocenici reperiti in grotte, specialmente in aree temperate a clima freddo.
H.N. Poinar e collaboratori (1996) hanno notato che la maggior parte dei campioni usati nel loro studio per identificare una correlazione tra la racemizzazione degli amminoacidi e il recupero del DNA provengono da ambienti caratterizzati da clima freddo. È intuibile che in tale clima la decomposizione microbica e autolitica possa essere ritardata al punto da permettere ai resti di deidratarsi riducendo enormemente il danneggiamento. La bassa temperatura può spiegare, in parte, l'eccellente stato di conservazione delle mummie Qilakitsoq della Groenlandia (Nielsen et al., 1994) che, pur avendo un'età di 1500 anni, sono state ritrovate in ottime condizioni perché deposte in tombe aerate in cui la temperatura non superava mai i 5°C. Un'eccezione interessante alla regola che il freddo e una scarsa presenza di umidità nell'aria sono necessari per la conservazione del DNA antico è rappresentata dai resti umani risalenti a 7000 ÷ 8000 anni fa scoperti a Windover, sul versante occidentale della catena costiera atlantica della Florida (Hauswirth et al., 1994). Questa zona, originariamente una palude alberata, è stata utilizzata come luogo di sepoltura tra l'8000 e il 6900 a.C.; i defunti vi venivano seppelliti alla profondità di circa un metro. Da allora, le condizioni non si sono particolarmente modificate e ora l'area è costituita da una torbiera satura d'acqua. La particolare composizione chimica dell'acqua presente nel terreno ha reso possibile la conservazione del tessuto nervoso, per altro l'unico tessuto molle rimasto intatto da cui i ricercatori abbiano potuto isolare con successo il DNA. L'alta percentuale di calcio, magnesio, carbonati, solfiti e solfati, nonché l'ambiente anaerobio, hanno ritardato la decomposizione dei tessuti biologici e la crescita di microrganismi. Gli autori ritengono che il DNA mitocondriale isolato dal tessuto nervoso dei corpi di Windover dimostri come si possa utilizzare DNA antico da resti provenienti da zone umide per studi sulle popolazioni umane primitive. Nonostante ciò, molti ricercatori si sono mostrati riluttanti a iniziare lo studio del DNA di popolazioni umane preistoriche poiché ritengono che vi sia un'alta possibilità di contaminazione esterna. M. Stoneking (1995) ha proposto una soluzione ideale, in cui il DNA umano antico sia diverso da quello dei tecnici che lavorano in laboratorio e che, in futuro, si debba consentire di lavorare con DNA umano antico solo a persone caratterizzate da genotipi rari.
M. Richards e collaboratori (1995) hanno offerto alcuni suggerimenti su come individuare DNA contaminante e hanno discusso il modo per autenticare i risultati ottenuti dall'estrazione di DNA da resti scheletrici primitivi. Tra l'altro essi propongono di replicare gli stessi esperimenti in laboratori differenti, con lo scopo di evidenziare le possibili contaminazioni che si verificano in laboratorio.
Antiche ossa umane da cui è stato ottenuto DNA appartengono a due individui di una razza africana simile al Cro Magnon, provenienti da Tafaralt, in Marocco, e risalenti a 12.000 anni fa (Beraud-Colomb et al., 1995). Più precisamente, risalgono all'epipaleolitico, un periodo che rappresenta una transizione dalla condizione di cacciatore-raccoglitore del paleolitico a quella di pastore-coltivatore del neolitico. Il rinvenimento di sequenze di DNA mitocondriale da ossa tipo Neandertal, datate tra 30 mila e 300 mila anni fa, ha aiutato a inquadrare la questione se gli uomini di Neandertal fossero antenati degli umani, o una linea di discendenza che con gli umani si poteva incrociare, o invece una specie distinta che non contribuì a fornire materiale genetico per la specie umana. l risultati hanno mostrato che il Neandertal si estinse senza fornire DNA mitocondriale agli umani moderni (Krings et al., 1997). Tali ritrovamenti sono risultati, quindi, di straordinario interesse per comprendere l'evoluzione umana.
T.A. Brown e K.A. Brown (1994), in una loro rassegna sul DNA antico, discutono sui vari tipi di informazioni che si possono acquisire dall'interpretazione dei siti archeologici e affermano che il DNA umano antico può essere utilizzato sia per confermare dati già acquisiti che per fornire nuove informazioni. Per esempio, nel caso della determinazione del sesso, qualora sia disponibile solamente un unico osso, è possibile teoricamente effettuare una PCR mirata ad amplificare specifiche sequenze ripetute presenti nel cromosoma Y umano per vedere se il DNA proviene da un individuo di sesso maschile. Anche se questo metodo non garantisce il successo, sono stati ottenuti risultati promettenti nell'ottenimento di porzioni del gene dell'amelogenina, una proteina dello smalto dei denti, presente nella regione pseudoautosomica del cromosoma del sesso. Nell'articolo di T.A. Brown e K.A. Brown troviamo questo e altri esempi relativi all'utilizzazione del DNA antico per studiare le migrazioni preistoriche, lo sviluppo dell'agricoltura e altri aspetti più specificamente pertinenti l'archeologia.
Un' interessante modalità indiretta per studiare l' evoluzione delle prime comunità umane è quello di cercare e analizzare resti di DNA dei cereali primitivi vicino ad abitazioni umane. T.A. Brown e collaboratori (1993), ideatori del metodo, hanno messo in evidenza che il DNA si conserva in grani di frumento carbonizzati. Non è noto esattamente il modo in cui i grani modificati dal calore conservino il loro DNA ma l'informazione che deriva dal loro studio può essere usata per studiare lo sviluppo iniziale dell' agricoltura e la sua diffusione nelle civiltà antiche. Inoltre utilizzando ancora come fonte di informazione reperti antichi di vegetali commestibili, G. Hillmann e collaboratori (1993) hanno ipotizzato la possibilità di determinare la dieta di animali estinti, analizzando i resti vegetali presenti nelle feci. Un ulteriore metodo per determinare attività e abitudini alimentari dell'uomo primitivo consiste nell'analizzare sangue, tessuti e capelli rimasti su strumenti litici (Loy, 1993).
Un aspetto interessante nell'indagine sul DNA antico di origine umana riguarda lo studio delle malattie dell'uomo primitivo nel tentativo di determinare se queste possano aver causato l'estinzione di popolazioni o la migrazione forzata dalle località in cui si svilupparono epidemie come, per esempio, l'allontanamento da sorgenti d'acqua in cui proliferavano mosche vettrici di agenti patogeni. D. Ortner e collaboratori (1992) hanno messo in evidenza la possibilità di ottenere sequenze di DNA primitive indicanti infezioni batteri che o virali. Il grande vantaggio rappresentato dall'utilizzo di tecniche di indagine sul DNA risiede, infatti, nella possibilità di poter estendere la ricerca a malattie che non si manifestino macroscopicamente nei tessuti o che producano uno sviluppo anormale negli organi.
Vi è un discreto interesse dell'opinione pubblica per studi sul DNA umano come quelli che riguardano l'identificazione delle vittime di guerra del Vietnam (Holland et al., 1993), di figure storiche, come la famiglia Romanov (Gill et al., 1994), di scoperte spettacolari come quella dell'uomo di Similaun descritta da Handt e collaboratori nel 1994 e l'identificazione dei resti di criminali, come Josef Mengele (Jefferys et al., 1992).
Fossili conservati in copale e ambra
Il copale e l'ambra sono resine che hanno subito un processo di fossilizzazione o di maturazione. l processi di polimerizzazione e di maturazione iniziano appena la resina fuoriesce dai tessuti vegetali; i dettagli relativi a questo processo non sono noti, comunque, nel giro di pochi mesi, la resina si indurisce al punto da poter esser fratturata esercitando una certa pressione. Questo materiale resinoso, relativamente morbido, non maturo e ancora di recente formazione, è chiamato copale. Il copale è reperibile in varie parti del globo (Nuova Zelanda, Africa, America Meridionale Repubblica Dominicana, Australia) con un'età compresa tra alcuni mesi e migliaia di anni. Indurendosi, il copale raggiunge una temperatura di fusione di 350°C, con gradi di durezza che raggiungono un valore di 2 o 3 della scala Mohr e la sua superficie non viene intaccata neppure in seguito al trattamento con solventi organici, quali acetone o etere. A questo punto, la resina fossilizzata ha raggiunto lo stadio definito di ambra, la cui formazione richiede generalmente milioni di anni. Attualmente vi sono molte località nel mondo dove si possono reperire resina, copale e ambra derivanti da una sola specie arborea. In Nuova Zelanda, a North lsland, è possibile raccogliere la resina che fuoriesce dalla corteccia di una conifera, il kauri, Agathis australis. Scavando il terreno intorno all'albero si può ottenere del copale originato si dalla stessa pianta. A molti chilometri di distanza si trovano rocce sedimentarie con pezzi di ambra kauri inclusi datati 40 milioni di anni. l fossili inclusi nel copale e nell' ambra hanno caratteristiche uniche poiché gli organismi sono rimasti immediatamente isolati dall'ambiente circostante nel momento in cui sono stati avvolti dalla resina. Tuttavia insetti di piccoli dimensioni e parti di piante non vengono completamente immersi nella resina istantaneamente e, a maggior ragione, insetti di grandi dimensioni e ancora vivi possono morire dopo essere rimasti attaccati alla superficie della resina e, quindi, rimanere esposti agli agenti atmosferici. In queste condizioni, funghi e batteri invadono l'organismo morto iniziando un processo di distruzione delle biomolecole che si può completare in sole 48 ore in ambiente tropicale.
Occasionalmente, ulteriore resina potrà ricoprire il corpo dell'insetto insieme al micelio e agli sporangiofori che sono cresciuti nel frattempo sul cadavere. La presenza di una crescita batterica è più difficile da identificare per l'assenza di evidenze esterne; è per questo motivo che si deve prestare molta attenzione nel selezionare insetti inclusi nell'ambra per escludere campioni che abbiano subito un processo di decomposizione. Insetti e parti di piante che siano stati immersi completamente da una singola colata di resina, rappresentano il campione ideale da cui isolare DNA antico.
l fossili inclusi nella resina attraversano dapprima lo stadio di copale e poi quello di ambra e, pertanto, durante questo periodo, sono soggetti unicamente ai cambiamenti chimici e fisici che si verificano nella resina. Essi sono protetti dagli agenti esterni in contatto con la superficie dell'ambra e, in questo modo, la tafonomia dell'ambra sembra essere piuttosto differente da quella del fossile che vi è incluso.
Non si conoscono ancora gli effetti dei vari componenti chimici della resina sulla stabilizzazione dei tessuti animali. R.e. Cambie (1976) ha identificato alcuni dei componenti chimici della resina del kauri; tra questi, zuccheri quali il glucosio, il galattosio e l'arabinosio potrebbero sottrarre l'acqua dai campioni inclusi per semplice osmosi. Ciò potrebbe portare a un tipo di disidratazione inerte, vantaggiosa per la conservazione del DNA. Anche i terpeni, come tali o ossidati ad aldeidi, noti agenti di fissaggio, potrebbero intervenire nella conservazione dei tessuti. La resina è insolubile in acqua e così, appena disidratato, il tessuto non può più essere reidratato a meno di una rottura dell' ambra stessa.
Alcuni anni fa si è discusso molto sulla possibile permeabilità della matrice dell'ambra all'ossigeno e ad altre molecole presenti nell'atmosfera (Hopfenberg et al., 1988). Quando l'ambra viene polverizzata o tagliata in fette sottili pochi micron, è possibile registrare un coefficiente di diffusione che indica che, almeno in questo caso, l'ambra potrebbe assorbire propano. Tuttavia, non sono state effettuate misurazioni sui vari gas in grado di penetrare dalla superficie dell'ambra nella matrice per alcuni millimetri. L'ambra intatta è, infatti, costituita da legami crociati tra polimeri che sino a un certo punto ritardano il processo di diffusione. Vi sono, evidentemente, ancora diversi quesiti insoluti che riguardano la diffusibilità nell'ambra; in uno studio recente sulla diffusione dell'argo, G.P. Landis e L.W. Snee (1991) hanno concluso che la ritenzione di argo radioattivo nei campioni di ambra indica che non c'è un rapido scambio dei gas contenuti nell'ambra con l'aria attualmente presente. L'estremamente basso coefficiente di diffusione dell'argo misurato dagli autori suggerisce che le bollicine di aria che si osservano nell'ambra contengono aria 'antica'.
Estrazione di campioni di tessuto da organismi inclusi nell'ambra
In tutto il mondo vi è un certo numero di depositi di ambra fossilifera risalenti al terziario e al cretaceo e ognuno di essi contiene rappresentanti unici della flora e della fauna. Questi depositi e gli esempi di forme viventi che essi contengono sono stati precedentemente discussi (Poinar, 1992; 1994). Qui volgeremo l'attenzione sull'estrazione del DNA da tessuti provenienti da organismi inclusi in ambra e per i quali l'estrazione del DNA sia stata documentata.
Per i campioni inclusi nell'ambra vi sono problemi di accessibilità, connessi al modo in cui possono essere raggiunti minimizzando i danni portati dall'esterno, cioè evitando di rovinare fisicamente i tessuti e di introdurre contaminanti. Le procedure per la sterilizzazione della superficie esterna dell'ambra e per l'estrazione dei tessuti sono state già descritte da G.O. Poinar e collaboratori (1994). Le modalità adottate originariamente consistevano nell' aprire il frammento di ambra lungo le linee di frattura causate da un bagno in azoto liquido, a cui immediatamente faceva seguito l'applicazione di una soluzione salina fisiologica calda. Tuttavia, questo metodo provocava diversi danni al campione e si rese quindi necessario ricorrere a un metodo meno distruttivo rappresentato dall'uso di micro-trapani. Con questo procedimento, si pratica nell'ambra un foro di ridotte dimensioni in prossimità del campione incluso; quindi si introduce un ago attraverso il foro fino alle pareti del corpo dell'esemplare estraendone i tessuti disidratati direttamente tramite l'ago o iniettando un liquido idoneo all'interno della cavità del corpo in modo da reidratarne i tessuti per poi estrarli successivamente. Quest'ultima metodica, in particolare, riduce il rischio di contaminazione esterna e non danneggia le strutture esterne del corpo dell'animale.
DNA antico da batteri
l batteri sono ubiquitari, non ci si deve quindi sorprendere di reperirli anche all'interno dell'ambra; in essa possono essere presenti, oltre alle spore batteriche, anche spore fungine e polline. Molto probabilmente, la presenza di batteri è dovuta alla loro diffusione come ectobionti sulla superficie esterna di piante e animali o come ospiti all'interno di resti di organismi morti prima che questi siano stati completamente ricoperti dalla resina. Nel libro di G.O. Poinar (1992) è riportata, oltre ad altri esempi, un'illustrazione in cui si può riconoscere una cellula batterica all'interno della cavità corporea di un nematode incluso in ambra, ritrovato in Messico. V. Galippe (1920) tentò perfrno di isolare alcuni batteri che aveva osservato in frammenti di ambra ritrovati nel Mar Baltico e riferì di essere stato in grado di isolare e mettere in coltura diverse specie batteriche, tra cui una che sembrava appartenere al genere Bacillus. Tuttavia, nessuno di questi batteri venne identificato e, in assenza di ulteriori approfondimenti, questo lavoro venne presto dimenticato. T. Hamamoto e K. Horikoshi (1994) sono riusciti a identificare un batterio isolato in un frammento di ambra del Baltico, il cui DNA presenta il 99,6% di omologia con quello di ceppi attuali di B. subtilis; nello stesso anno R.I. Cano e collaboratori (1994) sono stati in grado di recuperare DNA di Bacillus proveniente dall'addome dell'ape Problebeia dominicana, inclusa in ambra proveniente dalla Repubblica Dominicana e datato 25 ÷ 40 milioni di anni. Quella tra Bacillus e api rappresenta l'unica associazione simbiotica conosciuta che coinvolga specie di Bacillus dei giorni nostri (Poinar, 1996). l batteri vengono trasportati con il raccolto dalle api e la loro funzione sembra associata alla produzione di antibiotici necessari alla preservazione di proteine presenti nel polline, evitandone la degradazione da parte di funghi o altre specie batteriche.
Recentemente, R.I. Cano e M.K. Borucki (1995) hanno isolato e posto in coltura una delle specie simbiotiche di Bacillus provenienti dall'addome dell'ape ritrovata in frammenti di ambra della Repubblica Dominicana. l profili enzimatici, biochimici e della struttura del DNA codificante l'rRNA l6S, relativi a queste specie di Bacillus, hanno mostrato un'alta affinità con attuali ceppi di Bacillus sphaericus. A questo risultato hanno comunque fatto seguito osservazioni critiche dal punto di vista tecnico (Priest, 1995).
DNA antico proveniente da invertebrati
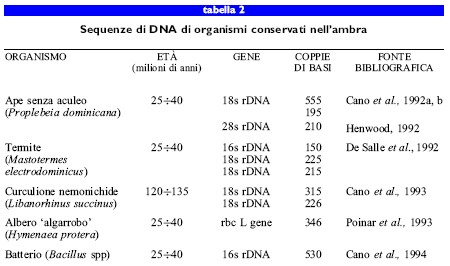
Uno studio relativo all'ultrastruttura del dittero Sciara coprophila incluso in un frammento di ambra del Mar Baltico, pubblicato nel 1982, diede un forte impulso alla ricerca del DNA antico di invertebrati conservati nell'ambra (Poinar e Hess, 1982). Da analisi ultrastrutturali si rilevò la presenza di nuclei integri dotati di cromatina nelle cellule epidermiche di questo dittero. Da allora, numerosi gruppi di ricercatori sono riusciti a ottenere DNA proveniente da insetti rimasti intrappolati nell'ambra (tab. 2).
La prima operazione di estrazione e di sequenziamento di un tratto di DNA ricavato da un organismo incluso in ambra è stata documentata da Cano e collaboratori (1992a; 1992b), che riferiscono di aver recuperato circa 500 bp relative al gene per l'rRNA l8S appartenente a una specie estinta di ape (Proplebeia dominicana) rinvenuta in un frammento di ambra proveniente dalla Repubblica Dominicana, risalente a 25 ÷ 40 milioni di anni fa.
Poco tempo dopo, R. De Salle e collaboratori (1992) furono in grado di estrarre e sequenziare circa 200 bp del medesimo gene e 150 bp del gene per l'rRNA l6S appartenenti a una termite (Mastotermes electrodominicus), ricavata da ambra della stessa provenienza della precedente. Nel corso di questo studio, si osservò il fenomeno del 'salto di PCR' (peR jumping) che consiste nel salto della polimerasi da uno stampo a un altro, durante la reazione di amplificazione e che porta alla formazione di molecole di DNA amplificato corrispondenti in parte alla sequenza desiderata e in parte a una sequenza contaminante (De Salle et al., 1993). Anche Hoelzel (Henwood, 1992) fu in grado di sequenziare 210 bp del gene per l'rRNA 28S dal DNA estratto dall'ape P. dominicana.
Il frammento di DNA più antico mai estratto finora da invertebrati preservati in ambra o in qualsiasi altro modo, corrisponde al tratto di un gene per l'rRNA l8S appartenente a un curculione (Libanorhinus succinus) con un'età di 120 ÷ 135 milioni di anni, estratto da ambra libanese del basso cretaceo (Cano et al., 1993). L'ambra libanese è, a tutt'oggi, quella che contiene i resti più antichi di insetti.
Vi è una certa preoccupazione relativa alla distruzione di specie rare di insetti inclusi nell'ambra per poterne prelevare il DNA. Si è già parlato di un metodo che consiste nel praticare un piccolo foro su un fianco dell'insetto, da cui possono essere prelevati campioni di tessuto per l'estrazione del DNA. Nel caso del raro curculionide è stato possibile ricomporre nuovamente i frammenti di ambra, in modo da poter studiare e descrivere le caratteristiche dell'insetto (Kuschel e Poinar, 1993).
L'analisi di frammenti di DNA antico risalenti a epoche così remote ha suscitato una certa discussione, soprattutto perché studi precedenti avevano mostrato che la degradazione del DNA si verifica, al massimo, nel corso di centomila anni (Lindahl, 1993a; 1993b). Se sarà possibile, in futuro, confermare gli studi sul DNA antico di organismi conservati in ambra e ripeterli in laboratori diversi, come sostengono A. Cooper e H.N. Poinar (1994), bisognerà concludere che l'effetto dovuto all'inclusione in una matrice di ambra determina un ritardo nella degradazione delle macromolecole. È già stata dimostrata la presenza di nuclei integri contenenti cromatina in organismi inclusi in ambra. Risultati ulteriori relativi alle modificazioni proteiche in organismi inclusi in ambra che intervengono nel corso del tempo sono coerenti all'affermazione che, in tali condizioni, il DNA risulta maggiormente preservato dalla degradazione. I primi studi sulla degradazione proteica effettuati su insetti inclusi in ambra (Bada et al., 1994) mostrarono che la racemizzazione degli amminoacidi è rallentata più di 100.000 volte rispetto alla norma. J.L. Bada e i suoi collaboratori scoprirono che gli amminoacidi di questi insetti sono molto simili a quelli delle forme attualmente conosciute e possono essere considerati come gli amminoacidi inalterati più antichi che si trovino sulla Terra. Uno studio successivo mostrò che, sebbene il contenuto totale di amminoacidi presente in insetti inclusi in ambra sia inferiore a quello di campioni attuali, sembra esservi un aumento di alcuni amminoacidi (Wang et al., 1996). Come già detto, H.N. Poinar e collaboratori (1996) hanno dimostrato che quando il rapporto D/L dell'acido aspartico in materiale fossile è 0,08 o meno c'è la possibilità di ottenere DNA antico dallo stesso campione e hanno anche trovato che il rapporto D/L dell'acido aspartico in cinque esemplari di insetti provenienti da copale e ambra variava da 0,01 a 0,08. Poiché il tasso di racemizzazione degli amminoacidi procede con un ritmo simile a quello del processo di depurinazione del DNA, gli studi sinora condotti rafforzano le conclusioni a cui erano precedentemente giunti Bada e collaboratori (1994), secondo cui anche la depurinazione del DNA racchiuso in frammenti di ambra subisce un rallentamento. Tutto ciò concorda con il recupero di sequenze di DNA da organismi inclusi in ambra, anche se è necessario attendere ulteriori prove da laboratori differenti.
DNA antico proveniente da piante conservate in ambra
Sino al momento della stesura di questo saggio è stato documentato solo un caso in cui è stato possibile estrarre DNA da una specie vegetale inclusa in ambra: nel 1993 H.N. Poinar e collaboratori riferirono dell'estrazione, amplificazione e sequenziamento di DNA, corrispondente al gene rbcL di cloroplasto, ricavato da una pianta estinta, la Hymenaea protera, racchiuso in un frammento di ambra rinvenuto nella Repubblica Dominicana. I dati molecolari ottenuti (Poinar et al., 1993) confermarono le precedenti conclusioni iniziali basate sulla morfologia, secondo cui la H protera della Repubblica Dominicana è filogeneticamente più vicina all'unica specie africana H verrucosa attualmente esistente, che non alle numerose specie di questo genere presenti nel Nuovo Mondo.
Un interessante aspetto collaterale è presente in uno studio di H.N. Poinar (1994), in cui si tentò di utilizzare i dati molecolari per stimare l'età dei depositi di ambra basandosi sul numero e sul tasso medio di sostituzioni nucleotidiche e sul numero di sostituzioni trovato per il gene rbcL. L'età, calcolata sulla base del tasso di sostituzione stimato da G. Zurawski e M.T. Clegg (1987) e per un totale di 34 sostituzioni, risultò essere di 38,4 milioni di anni.
Sebbene questo metodo di calcolo dell'età dei reperti sia ancora altamente congetturale, il dato ottenuto concordava pienamente con stime precedenti effettuate su frammenti di ambra rinvenuti nella miniera di La Toca, nella Repubblica Dominicana.
DNA antico proveniente da resti di piante
In questo paragrafo prenderemo in esame i risultati relativi all'estrazione di DNA da resti di piante non rinvenuti in ambra.
Le foglie e i semi costituiscono i due tessuti principalmente utilizzati per il recupero di DNA antico di piante. Inizialmente, si sospettava che il destino del DNA appartenente a piante arcaiche fosse differente da quello degli animali, essendo il primo protetto da una parete di cellulosa. Nel 1988, F. Rollo e collaboratori (Rollo et al., 1988) fornirono la prima prova della presenza di DNA endogeno in semi di piante arcaiche. In una rassegna sugli studi effettuati su questo argomento, Rollo e collaboratori (1994a) presentarono le procedure di estrazione e discussero le problematiche relative alle contaminazioni da microbi. Gli autori posero espressamente l'accento su quest'ultimo aspetto, in relazione all'utilizzo di semi preservati per estrarre DNA e sulla conseguente necessità di operare con precauzione, come nel corso della manipolazione di resti animali. Nella pubblicazione in cui presentavano l'analisi molecolare di una pianta di 5000 anni fa, rinvenuta assieme all'uomo di Similaun, Rollo e collaboratori (1994b) riferirono del recupero di una gran quantità di DNA non appartenente alla pianta, ma a funghi e alghe, probabilmente associati alla pianta.
Nella scelta della sequenza di DNA da analizzare, Rollo e collaboratori fecero notare come, pur essendo il DNA mitocondriale una possibile scelta per queste analisi, il suo tasso di mutazioni puntiformi risulta, nelle piante, 100 volte più lento che negli animali. D'altronde, il DNA dei cloroplasti, pur essendo particolarmente abbondante nei tessuti foto sintetici, rappresenta solo l'1% del DNA totale contenuto in un seme. Infine, poiché il DNA nucleare delle piante evolve a una velocità doppia rispetto a quella del DNA dei cloroplasti, il DNA nucleare rappresenta la migliore fonte di DNA per questo tipo di analisi.
Nel 1990, il mondo scientifico fu sorpreso dalla notizia che il DNA si può conservare nei tessuti delle piante per milioni di anni. E.M. Golenberg e collaboratori (1990) riferirono sul recupero di circa 820 bp, appartenenti al gene rbcL di cloroplasto, da una foglia di magnolia datata 17 ÷ 20 milioni di anni, rinvenuta nei depositi di Clarkia nel Nord dell'ldaho. Questo costituì il primo risultato relativo al recupero di DNA antico di più di un milione di anni. Le foglie si erano conservate racchiuse in strati di argilla morbida, finemente laminata e non ossidata. Questi strati di argilla costituiscono il fondo di un lago miocenico formatosi rapidamente a seguito di un'eruzione vulcanica, avvenuta nelle vicinanze, che ostruì il letto di un ruscello. Le foglie si erano accumulate sul fondo del lago, dove vi erano probabihnente condizioni di anossia. l tessuti vegetali erano ancora non ossidati e impregnati di acqua al momento della raccolta per l'estrazione del DNA. S. Pääbo e A.C. Wilson (1991) determinarono che in tali condizioni ambientali, a 15°C di temperatura e un valore di pH pari a 7, un frammento di DNA costituito da 800 bp si depurinirebbe completamente in circa 5000 anni. Questi ultimi autori hanno anche tentato di ricavare DNA da altre foglie scavate successivamente e benché verificassero che un estratto su dieci conteneva DNA, trovarono che questo aveva un'origine batterica anziché provenire da piante superiori (Sidow et al., 1991). Conclusero così che sarebbero state necessarie ulteriori ricerche per verificare questi risultati.
Nel commentare questi fatti, Golenberg (1991) suggerì che alcune trasformazioni intervenute nei tessuti delle foglie provenienti dai depositi di Clarkia potevano spiegare la mancata depurinazione del DNA dopo 20 milioni di anni. Una possibilità è che le foglie fossero state sottoposte a un processo naturale di disidratazione prima di cadere dall'albero e ciò avrebbe provocato la rottura dei tonoplasti e il rilascio di fenoli che, normalmente, vengono prodotti da una pianta.
Nel 1992 un altro gruppo di ricercatori riferì di aver estratto, amplificato e sequenziato una porzione di 1320 bp del gene rbcL del cloroplasto di Taxodium (un cipresso caducifoglio), proveniente dagli stessi depositi di Clarkia (Soltis et al., 1992), avvalorando così i risultati precedentemente ottenuti. Le foglie provenienti dai depositi di Clarkia vennero anche analizzate in relazione alla racemizzazione degli amminoacidi da H.N. Poinar e collaboratori (1996); sebbene il quantitativo di acido aspartico presente nei quattro campioni di foglie analizzati fosse troppo basso per consentire una misurazione dei prodotti di racemizzazione, fu comunque possibile rilevare la presenza di alanina destrogira e levo gira e il rapporto di racemizzazione D/L risultò uguale o maggiore a 0,15. Gli autori notarono come, di fronte a valori così alti, non ci si può aspettare una buona conservazione del DNA.
Ovviamente, non è stato possibile rilevare la presenza di DNA in tutti i campioni provenienti dallo stesso habitat, che sono stati analizzati negli studi menzionati. La soluzione di risultati contrastanti, come quelli relativi agli studi effettuati sui depositi di Clarkia, rappresenta una delle sfide più affascinanti per i ricercatori che agiscono nel campo del DNA antico.
Sviluppi futuri
Oltre agli articoli che continuamente riferiscono dei progressi compiuti in materia di estrazione e analisi del DNA proveniente da fossili e che sono stati presentati in questo saggio, esistono altre aree di interesse generale ancora da esplorare, tra cui lo studio delle malattie del passato non solo dell'uomo ma anche degli altri vertebrati, degli invertebrati e delle piante. Questo genere di studi può essere affrontato mediante l'isolamento di sequenze di DNA appartenute ad agenti patogeni individuati direttamente nel loro ospite definitivo, che può essere rappresentato da un vertebrato, un invertebrato o una pianta. Altrimenti, nel caso di malattie che si propagano mediante un vettore, si può tentare un approccio indiretto basato sull'isolamento di sequenze di DNA del patogeno dall'ospite intermedio che è generalmente un invertebrato nel caso delle malattie di vertebrati, come la malaria, l'infezione da arbovirus, la filariosi, la malattia di Lyme, la schistosomiasi e molte altre.
l numerosi progetti volti a definire la mappa genetica di animali, utilizzati specificamente come sistemi-modello, come la Drosophila, dovrebbero consentire, una volta che si siano rese disponibili singole sequenze o gruppi di sequenze responsabili del controllo o della modificazione di determinati caratteri, di introdurre in ceppi di laboratorio sequenze ricavate da drosofilidi fossili conservati in ambra, in modo da ottenere l'espressione di caratteri appartenuti a specie estinte.
Con l'avanzare delle conoscenze sulla struttura molecolare delle piante, sarà possibile ricercare in specie fossili, oggi estinte, sequenze geniche in grado di conferire resistenza a determinate malattie. Tali sequenze potrebbero essere inserite in piante coltivate, determinando una protezione nei confronti di un ampio spettro di organismi patogeni e traendo così notevoli vantaggi per l'agricoltura.
Conclusioni
Gli studiosi del DNA antico sono giunti al punto di riconsiderare e riflettere sul grande numero di dati raccolti negli anni Novanta. l dati vengono sottoposti a scrupolose verifiche eseguite in laboratori differenti, in modo da poter essere confermati o smentiti. Questa serie di verifiche si è resa necessaria in seguito alla scoperta delle numerose forme di contaminazione che si possono verificare, ai possibili errori durante la reazione di PCR e alla diffusione di risultati sulla longevità del DNA antico. Alla luce di queste nuove informazioni, i ricercatori che operano sull'argomento procedono oggi con molta prudenza, in quanto si è compreso come si debba tener conto di un numero di variabili più alto di quanto si pensasse quando si è iniziato a operare con il DNA antico. Non c'è dubbio che la ricerca nel campo del DNA antico rivesta una notevole importanza, costituendo un valido strumento d'indagine nello studio degli organismi estinti, delle loro filogenesi, migrazioni e distribuzioni. Sarà interessante vedere se i risultati delle analisi di tipo molecolare confermeranno o meno il punto di vista di precedenti ricerche che basavano le loro conclusioni su metodi di analisi più tradizionali.
Bibliografia citata
BADA, J.L., W ANG, X.S., POINAR, H.N., PÄÄBO, S., POINAR, G.O. (1994) Amino acid racemization in amber-entombed insects: Implication for DNA preservation. Geochimica Cosmochimica Acta, 58, 3131-3135.
BERAUD-COLOMB, E., ROUBIN, R., MARTIN, J., MAROC, N., GARDEISEN, A., TRABUCHET, G., GOOSSENS, M. (1995) Human beta-globin gene polymorphisms characterized in DNA extracted from ancient bones 12,000 years old. Am. J. Rum. Genet., 57, 1267-1274.
BROWN, T.A., ALLABY, G.A., BROWN, K.A., JONES, M.K. (1993) World Archaeology, 25, 64-73.
BROWN, T.A., BROWN, K.A. (1994) Ancient DNA: using molecular biology to explore the past. Bioessays, 16,719-726.
CAMBIE, R.C. (1976)J. Royal Society of New Zealand, 342, 307-379.
CANO, R.J., BORUCKI, M.K. (1995) Revival and identification of bacterial spores in 25-to 40-million-year-old Dominican amber. Science, 268, 1060-1064.
CANO, R.J., BORUCKI, M.K., HIGBY-SCHWEITZER, M., POINAR, H.N., POINAR, G.O., POLLARD, K.I. (1994) Bacillus DNA in fossil bees: an ancient symbiosis? Appl. Environ. Microbiol., 60, 2164-2167.
CANO, R.I., POINAR, H.N., PIENIAZEK, N.I., ACRA, A., POINAR, G.O.Jr.(1993) Amplification and sequencing of DNA from a 120-135 million-year old weevil. Nature, 363, 536-538.
CANO, R.J., POINAR, H.N., POINAR, G.O. (1992a) Isolation and partial characterization of DNA from the bee Proplebeia dominicana (Apidae: Rymenoptera) in 25-40 million year old amber. Med. Sci. Res., 20, 249-251.
CANO, R.J.., POINAR, H.N., POINAR, G.O. (1992b) Enzymatic amplification and nucleotide sequencing of portions of the 18S rRNA gene of the bee Proplebeia dominicana as isolated from 25-40 million year amber. Med. Sci. Res., 20, 619-622.
COOPER, A., MOURER-CHAUVIRE, C., CHAMBERS, G.K., VON HAESELER, A., WILSON, A.C., PÄÄBO, S. (1992) Independent origins of New Zealand moas and kiwis. Proc. Natl. Acad. Sci., 89, 8741-8744.
COOPER, A., POINAR, H.N. (1994) Contamination and negative results in ancient DNA studies and amber reserch. Ancient DNA NewsIetter, 2, 6-7.
DE SALLE, R., GATESY, J., WHEELER, W., GRIMALDI, D. (1992) DNA sequences from a fossile termite in Oligo-Miocene amber and their phylogenetic implications. Science, 257,1933-1936.
DE SALLE, R., BARCIA, M., WRAY, C. (1993) PCR jumping in clones of 30-million-year-old DNA fragments from amber preserved termites (Mastotermes eIectrodominicus). Experientia, 49, 906-909.
GALIPPE, V. (1920) Recherches sur la resistance des microzymas a l'action du temp et sur leur survivance dans l'ambre. Comp. Rendu Acad. Sci. (Parigi), 170, 856-858.
GEIGER, R. (1959) The climate near the ground. Cambridge, Harvard University Press.
GILL, P., IVANOV, P.L., KiMPTON, C., PIERCY, R., BENSON, N., TULLY, G., EVETT, I., HAGELBERG, E., SULLIVAN, K. (1994) Identification of the remains of the Romanov family by DNA analysis. Nat. Genet., 6, 130-135.
GOLENBERG, E.M. (1991) Amplification and analysis of Miocene plant fossil DNA. Phil. Trans. R. Soc. Lond. B, 333, 419-426.
GOLENBERG, E.M., GIANNASI, D.E., CLEGG, M.T., SMILEY, C.I., DURBIN, M., HENDERSON, D., ZURAWSKI, G. (1990) ChIoroplast DNA sequence from a Miocene Magnolia species. Nature, 344, 656-658.
HAGELBERG, E. (1994) Ancient DNA. New York, Springer-Verlag.
HAGELBERG, E., SYKES, B., HEDGES, R. (1989) Ancient bone DNA amplified. Nature, 342, 485.
HAGELBERG, E., THOMAS, M.G., COOK, C.E.Jr., SHER, A.V., BARYSHNIKOV, G.F., LISTER, A.M. (1994) DNA from ancient mammoth bones. Nature, 370, 333-334.
HAMAMOTO, T., HORIKOSHI, K. (1994) Characterization of a bacterium isolated from amber. Biodiversity and Conservation, 3, 567-572.
HANDT, O., HÖSS, M., KRINGS, M., PÄÄBO, S. (1994) Ancient DNA: methodological challenges. Experientia, 50, 524-529.
HANDT, O., et al. (1994) Molecular genetic analyses of the Tyrolean ice mano Science, 264, 1775-1778.
HAUSWIRTH, W.W., DICKEL, C.D., LAWLOR, D.A. (1994) In Ancient DNA. New York Springer-Verlag.
HEDGES, S.B., SCHWEITZER, M.H. (1995) Detecting dinosaur DNA. Science, 268, 1191-1192.
HENWOOD, A.A. (1992) Insect taphonomy from Tertiary amber of the Dominican Republic. Ph.D. Dissertation, University of Cambridge, Gran Bretagna.
HIGUCHI, R., BOWMAN, B., FREIBERGER, M., RYDER, O.A., WILSON, A.C. (1984) DNA sequences from the quagga, an extinct member of the horse family. Nature, 312, 282-284.
HILLMAN, G., WALES, S., MCLAREN, F., EVANS, J., BUTLER, A. (1993) Identifyng problematic remains of ancient plant foods: a comparison of the role of chemical, histological and morfological criteria. World Archaeology, 25, 94-121.
HOLLAND, M.M., FISHER, D.L., MITCHELL, L.G., RODRIQUEZ, W.C., CANIK, J.J., MERRIL, C.R., WEEDN, V.W. (1993) Mitochondrial DNA sequence analysis of human skeletal remains: identification of remains from the Vietnam war. ] Forensic Sci., 38, 542-553.
HOPFENBERG, H.B., WITCHEY, L.C., POINAR, G.O. (1988) Is the air in amber ancient? Science, 241, 717-718.
HORAI, S., HAYASAKA, K., WATE, N., KOIKE, H., NAKAI, N. (1989) DNA amplification from ancient human skeletal remains and their sequence analysis. Proc. Jap. Academy, 65, 229-233.
HÖSS, M., DILLING, A., CURRANT, A., PÄÄBO, S. (1996) Molecular phylogeny ofthe extinct ground slothMyIodon darwinii. ProC. Natl. Acad. Sci., 92, 181-185.
HÖSS, M., PÄÄBO, S., VERESHCHAGIN, N.K. (1994) Mammoth DNA sequences. Nature, 370, 333.
JANCZEWSKI, D.N., YUHKI, N., GILBERT, D.A., JEFFERSON, G.T., O'BRIEN, S.J. (1992) Molecular phylogenetic inference from saber-toothed cat fossils of Rancho La Brea. Proc. Natl. Acad. Sci., 89, 9769-9773.
JEFFREYS, A.J., ALLEN, M.J., HAGELBERG, E., SONNBERG, A. (1992) Identification of the skeletal remains of Josef Mengele by DNA analysis. Forensic Sci. Int., 56, 65-76.
KRAJEWSKI, C., DRISKELL, A.C., BABERSTOCK, P.R., BRAUN, M.I. (1992) Phylogenetic relationships of the thylacine (Mammalia: Thylacinidae) among marsupials: from evidence cytochrome bDNA sequences. Proc. Roy. Soc. London B, 250, 19-27.
KRINGS, M., STONE A., SCHMITZ, R.W., KRAINITZKI, H., STONEKING, M., PÄÄBO, S. (1997) Neandertal DNA sequences and the origin of modem humans. CelI, 90, 19-30.
KUSCHEL, G., POINAR, G.O.Jr., (1993) Lebanorhius succinus gen. et sp. n. (Coleoptera: Nemonychoidae) from Lebanese amber. Ent. scand., 24, 143-146.
LANDIS, G.P., SNEE, L.W. (1991) Ar/Ar systematics and argon diffusion in amber: implications for ancient earth atmospheres. PaIaeogeography, PaIaeoclimatology, PaIaeoecology, 97, 6367.
LEWIN, P.K. (1967) Palaeo-electron microscopy of mummified tissue. Nature, 213, 416-417.
LINDAHL, T. (1993a) Instability and decay ofthe primary structure of DNA. Nature, 362, 709-715.
LINDAHL, T. (1993b) Recovery of antediluvian DNA. Nature, 365, 700.
LOY, T.H. (1993) The artifact as site: an example ofthe biomolecular analysis of organic residues on prehistoric tools. World Archaeology, 25, 44-63.
MANEN, J.-F., SAVOLAINEN, V., DE MARCHIN, S., RION, B. (1995) CR. Acad. Sci. Paris, sciences de la vie, 318, 971-975.
NIELSEN, H., ENGBERG, J., THUESEN, I. (1994) DNA from arctic Human burials. In Ancient DNA, a c. di Herrmann B.C., Hummel S., New York, Springer-Verlag, pp. 122-140.
ORTNER, D.l., TUROSS, N., STIX, A.I. (1992) New approaches to the study of disease in archeological New World populations. Rum. biol., 64, 337-360.
PÄÄBO, S. (1985) Molecular cloning of ancient Egyptian mummy DNA. Nature, 314, 644-645.
PÄÄBO, S. (1986) Molecular genetic investigations of ancient human remains. Cold Spring Rarb Symp. Quant. Biol., 51, 441-446.
PÄÄBO, S. (1989) Ancient DNA: extraction, characterization, molecular cloning, and enzymatic amplification. Proc. Natl. Acad. Sci., 86, 1939-1943.
PÄÄBO, S. (1990) in PCR Protocols. New York, Academic Press, pp. 159-166.
PÄÄBO, S. (1991) Amplifying DNA from archeological remains: a meeting report. PCR Methods Appl., 1, 107-110.
PÄÄBO, S., Wilson, A.C. (1991) Miocene DNA sequences: a dream come true? Current Biology, 1, 45-46.
POINAR, G.O.Jr. (1992) Life in amber, Stanford, Stanford University Press.
POINAR, G.O.Jr. (1994) The range of life in amber: significance and implications in DNA studies. Experientia, 50, 536-542.
POINAR, G.O.Jr. (1996) Entomopathogenic Manual, Corporacion para Investigaciones Biologicas, in stampa.
POINAR, G.O.Jr., HESS, R. (1982) Ultrastructure of 40-millionyear-old insect tissue. Science, 215, 1241-1242.
POINAR, G.O.Jr., POINAR, H.N., CANO, R.I. (1994) DNA from amber inclusions. In Ancient DNA, a C. di Herrmann B.C., Hummel S., New York, Springer-Verlag, pp. 92-103.
POINAR, H.N. (1994) Molecular phyIogeny and biogeography of an extinct Iegume (Rymenaea protera) from Dominican amber. San Luis Obispo, M.S. dissertation, California Polytechnique State University.
POINAR, H.N., HESS, M., BADA, J.L., PÄÄBO, S. (1996) Origins and evolution of Bacillus in relation to insect parasitism. Science, 272, 864-866.
POINAR, H.N., POINAR, G.O.Jr., CANO, R.I. (1993) DNA from extinct plant. Nature, 363, 677.
PRIEST, F.G. (1995) Age of bacteria from amber. Science, 270, 2015.
RICHARDS, M.B., SYKES, B.C., HEDGES, R.E.M. (1995) Authenticating DNA extracted from ancient skeletal remains. J. Archaeological Science, 22, 291-299.
RICHARDSON, S. (1994) Discover, 15, 55.
ROLLO, F., AMICI, A., SALVI, R., GARBUGLIA, A. (1988) Short but faithful pieces of ancient DNA. Nature, 335, 774.
ROLLO, F., ASCI, W., ANTONINI, A., MAROTA, I., UBALDI, M. (1994b) Molecolar ecology of a Neolithic meadow: the DNA of the grass remains from the archaeological site of the Tyrolean Iceman. Experientia, 50, 576-584.
ROLLO, F., VENANZI, F.M., AMICI, A. (1994a) DNA and RNA from ancient plant seeds. In Ancient DNA, a C. di Herrmann B., Hummel S., New York, Springer-Verlag, pp. 218-236.
SIDOW, A., WILSON, A.C., PÄÄBO, S. (1991) Bacterial DNA in CIarkia fossils. Trends Genet., 7, 4.
SMITH, K.G. (1986) A manual of Jorensic entomology. British Museum of Natural History, Ithaca, Cornell University Press.
SOLTIS, P.S., SOLTIS, D.E., SMILEY, C.J. (1992) An rbcL sequence from Miocene Taxodium (bald cypress). Proc. Natl. Acad. Sci., 89, 449-451.
STONEKING, M. (1995) Ancient DNA: how do you know when you have it and what can you do with it? Am. J. Rum. Genet., 57, 1259-1262.
WANG, X.W., BADA, J.L., POINAR, H.N., POINAR, G.O.Jr. (1996) Amber, Resinite and Fossil Resin, ACS symposium series 617, American Chemical Society, Washington, D.C., pp. 225-262.
WAYNE, R., COOPER, A. (1992) Ancient DNA NewsIetter. Institute of Zoology, Zoological Society of London, 1, 1-42. WOODWARD, S.R., WEYAND, N.J., BUNNELL, M. (1994) DNA sequence from Cretaceous period bone fragments. Science, 266, 1229-1230.
ZISCHLER, H., HÖSS, M., VON HAESELER, A., VAN DER KUYL, A.C., GOUDSMIT, J., PÄÄBO, S. (1995) Detecting dinosaur DNA. Science, 268, 1192-1193.
ZURAWSKI, G., CLEGG, M.T. (1987) Ann. Rev. Plant Physiol., 38, 391-418.
Bibliografia generale
AVISE, J.C. Molecular markers, natural history and evolution. New York, Chapman and Hall, 1994.
BROWN, T.A., BROWN, K.A. (1992) Antiquity, 66, 10-23.
DARWIN, C. The Voyage of the Beagle. Garden City, New York, Doubleday & Co., Inc., 1860.
EXPERIENTIA (1994) Ancient DNA., 50, 521-601.
HERMANN, B., HUMMEL, S. a c. di Ancient DNA, New York, Springer-Verlag, 1994.
LEETON, P., CHRISTIDIS, L. (1993) Feathers from museum bird skins- a good source of DNA for phytogenetic studies. The Condor, 95, 465-466.
PÄÄBO, S. (1993) Ancient DNA. Sci Am, 269, 86-92.
ROSS, P.E. (1992) Eloquent remains. Sci Am, 266, 114-119.
SOLTIS, P., SOLTIS, D.E. (1993) Ancient DNA: prospects and limitations. New Zealand J. of Botany, 31, 203-209.
THOMAS, K.D. (1993) Molecular biology and archeology: a prospectus for inter-disciplinary research. World Archaeol., 25, 1-17.
WAYNE, R.K. (1993) Molecular evolution of the dog family. Trends Genet., 9, 218-224.
© Istituto della Enciclopedia Italiana - Riproduzione riservata